Partager la publication "Covid 19 : Calcul du nombre de jours en réanimation par département"
Dans cet article, nous verrons comment déterminer un nombre de jours en service de réanimation par département à partir des données de Santé Publique France, au moyen d’un programme Python.
Normalement, si l’on voulait calculer ce nombre de jours de façon certaine, il faudrait avoir un fichier de toutes les observations (c’est à dire dans notre cas de toutes les hospitalisations).
Ce fichier devrait contenir des informations sur chaque personne sur la date d’hospitalisation, la date d’entrée en réanimation, la date de sortie de réanimation et selon les cas la date de décès ou de retour à domicile.
Voir un exemple ci-dessous :
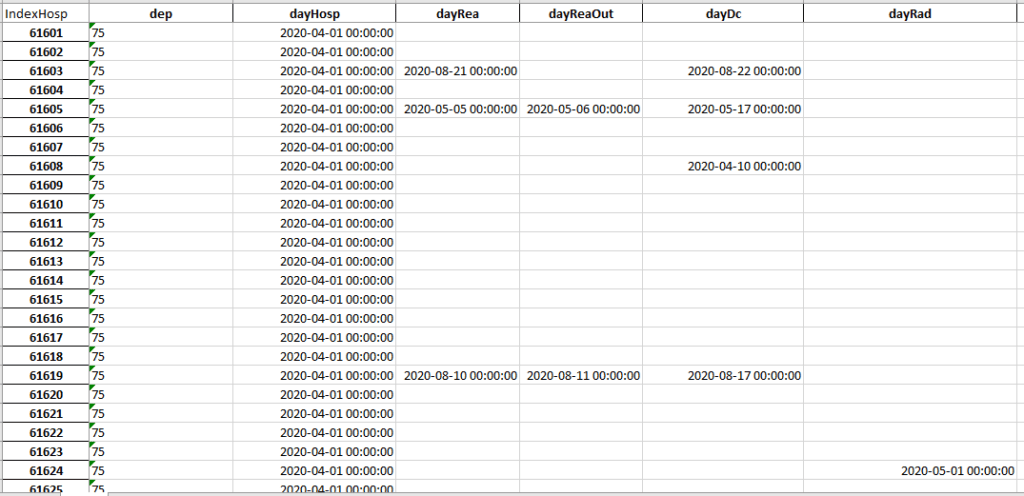
Ces données n’étant pas disponibles en l’état, nous allons être obligés d’utiliser le hasard pour créer celles-ci .
Nous procéderons donc au tirage de plusieurs échantillons (par exemple 100 mais 20 peuvent suffire) pour dégager des moyennes de durée par département.
Le fichier des observations sur les hospitalisations sera lui-même construit à partir des données de nouvelles hospitalisations, nouvelles admissions en réanimation, nouveaux retours à domiciles et nouveaux décès par département et par jour.
Soit les données d’incidences par jour et par département ici en jaune :
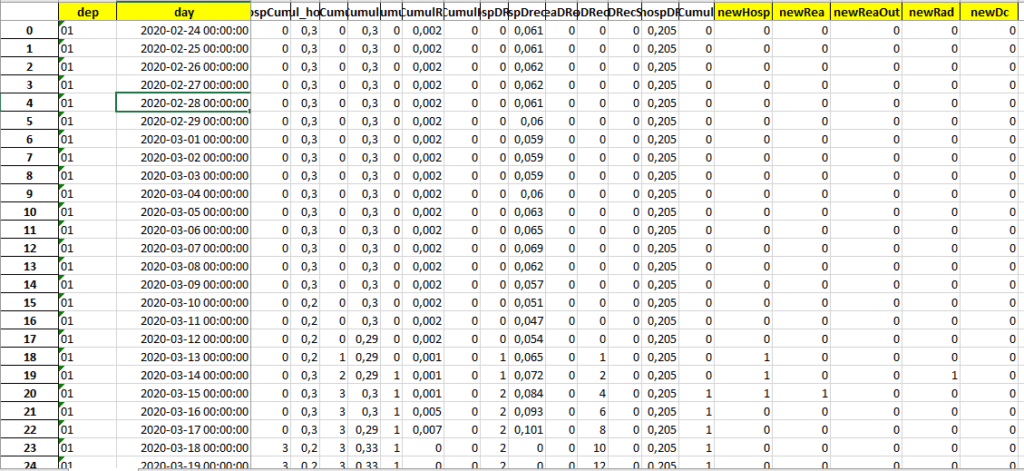
Ces données n’étant pas non plus disponibles dans les fichiers de Santé Publique France elles seront aussi reconstruites.
De quoi aurons nous besoin ?
Python
Nous conseillons d’utiliser la version de Python Anaconda qui contient déjà toutes les bibliothèques dont nous pourrions avoir besoin.
Pour cela, allez à l’adresse https://www.anaconda.com/products/individual pour télécharger la dernière version qui vous convient :

Anaconda est fourni avec l’Environnement de Développement Intégré (IDE : Integrated Development Environment) Spyder :
Nous ne détaillons pas ici l’utilisation de Python Anaconda que nous avons déjà abordée par ailleurs.
Reportez-vous à la documentation de l’éditeur https://docs.anaconda.com/anaconda/
Données Santé Publique France
Pour ce programme nous allons utiliser les données de Santé publique France contenues dans 2 types de fichiers :
Fichier « Données hospitalières »
Le fichier des données hospitalières est selon nous le plus « fiable » car c’est celui qui donne le nombre de décès par jour. Cette donnée est à notre avis la donnée la mieux répertoriée par les hôpitaux.
Les fichiers de ce type sont sous le nom « donnees-hospitalieres-covid19-YYYY-MM-DD-19h00.csv » et sont récupérables sur le site de Data.gouv.fr à l’adresse https://www.data.gouv.fr/en/datasets/donnees-hospitalieres-relatives-a-lepidemie-de-covid-19/
Voici la structure de ces types de fichiers, en jaune les données qui nous intéressent :
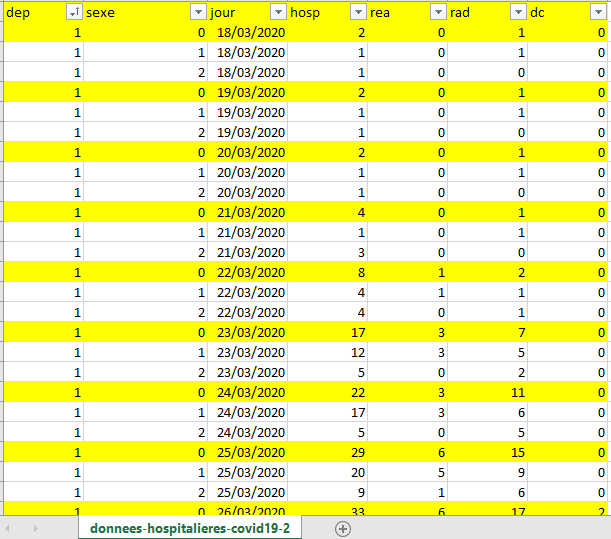
Pour cet exercice, nous n’utiliserons pas les données hommes/femmes et ne garderons que les données agrégées.
Notez bien que les données hosp et rea correspondent aux nombres d’hospitalisations le jour J et que les données rad (Retour à domicile) et dc (Dècès) sont des cumuls depuis le début de l’épidémie.
Pour la suite, il sera utile de créer des cumuls pour toutes les variables pour en déduire le nombre d’incidences (nouveaux cas) par jour.
Vous pouvez aussi noter que ce fichier est mal « rangé » : les données pour les hommes et les femmes auraient du se trouver sur la même ligne que le global selon le principe d’une observation par jour en ligne et les variables en colonnes.
Enfin, vous pouvez constater que les données démarrent le 18/03/2020 alors que l’épidémie a démarré bien avant.
Pour certains départements les données du 18/03/2020 se retrouvent difficilement exploitables.
C’est pourquoi nous allons aussi utiliser les données des urgences :
Fichier « Sursaud »
Les fichiers « sursaud » contiennent des informations sur l’activité des urgences et de SOS Médecins et liées au Covid19.
Dans ce cas on ne s’intéressera pas aux données de SOS Médecins.
Les fichiers de ce type sont sous le nom « sursaud-corona-quot-dep-YYYY-MM-DD-19h15.csv » et sont récupérables sur le site de Data.gouv.fr à l’adresse https://www.data.gouv.fr/en/datasets/donnees-des-urgences-hospitalieres-et-de-sos-medecins-relatives-a-lepidemie-de-covid-19/
Voici la structure de ces types de fichiers, en jaune les données qui nous intéressent :
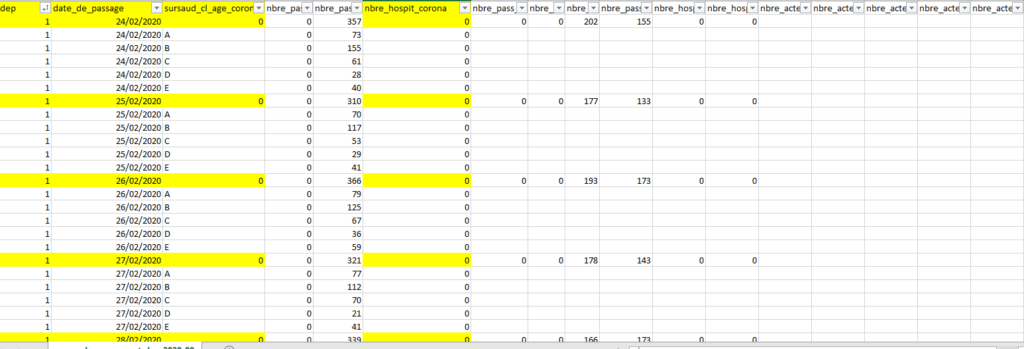
On ne s’intéressera pas aux données de classes d’âge et on récupérera uniquement le nombre d’hospitalisations parmi les passages aux urgences pour suspicion de COVID-19 : nbre_hospit_corona. Notez que ce nombre est une incidence.
Notez aussi que les données démarrent à partir du 24/02/2020, ce qui permet d’avoir des données assez tôt dans l’épidémie. On ne perd que quelque cas.
Le défi de la première partie du programme consistera à reconstruire des données cohérentes sur la période du 24/02/2020 au 18/03/2020.
Code Source
Vous pouvez télécharger le code source en entier dans notre boutique à l’adresse https://www.anakeyn.com/boutique/produit/covid19-jours-rea-dep/
Sinon vous pouvez copier/coller les morceaux de code 1 par 1
Chargement des bibliothèques utiles et des noms de fichiers de Santé Publique France
# -*- coding: utf-8 -*- """ Created on Mon Sep 13 16:28:24 2020 @author: Pierre """ import pandas as pd import numpy as np from datetime import datetime #, timedelta, date import random import statistics #for mean #Define files with original data sursaudFile = "sursaud-corona-quot-dep-2020-09-14-19h15.csv" hospFile = "donnees-hospitalieres-covid19-2020-09-14-19h00.csv"
Notez que les fichiers doivent se trouver dans le répertoire de travail défini en haut à droite dans Spyder.
Partie I : création des données d’incidences : newHosp, newRea, newReaOut, newRad, newDc à partir des données de Santé Public France.
Import des données « sursaud » et création d’une variable de cumul des hospitalisations suite aux urgences « cumul_hospit_corona »
######################################################################################
# I - create newHosp, newRea, newReaOut, newRad, newDc Tidy data dataframe/file
######################################################################################
###############################################################
# Read sursaud file :
# i.e sursaud-corona-quot-dep-YYYY-MM-DD-19h15.csv
###############################################################
#Import file with necessary variables
dateparse = lambda x: pd.to_datetime(x, format='%d/%m/%Y')
df = pd.read_csv(sursaudFile, sep=";" ,
usecols= ["dep","date_de_passage","sursaud_cl_age_corona", "nbre_hospit_corona"],
dtype={"dep": str}, #force dep to string
parse_dates= ["date_de_passage"], date_parser=dateparse)
df.dtypes
df.rename(columns={"date_de_passage": "day" }, inplace=True)
df["nbre_hospit_corona"] = df["nbre_hospit_corona"].fillna(0) #replace NaN by 0
#df['dep'].astype(str) #force dep to string #done in import
df['dep'] = df['dep'].str.strip() #strip white spaces
#select only global data
df = df.loc[df['sursaud_cl_age_corona'] == '0']
#remove age classe column
dfUrgences = df.drop(columns=['sursaud_cl_age_corona'])
#Reindex
dfUrgences.reset_index(inplace=True, drop=True) #reset index
#add cumul column
dfUrgences['cumul_hospit_corona']=0.0
dfUrgences.dtypes
previousDep = "00"
previousCumul = 0.0
#calculate cumul_hospit_Corona
for index, row in dfUrgences.iterrows():
print("index:", index)
print("dep:", row['dep'])
print("date:", row['day'])
if (index==0) | (row['dep']!=previousDep):
previousCumul = 0.0
dfUrgences.loc[index, 'cumul_hospit_corona'] = row['nbre_hospit_corona']
print("First line")
else :
dfUrgences.loc[index, 'cumul_hospit_corona'] = row['nbre_hospit_corona'] + previousCumul
print("other lines")
previousDep = row['dep']
previousCumul = row['nbre_hospit_corona'] + previousCumul
print("previousCumul:", previousCumul)
#Save Urgences data if needed
#dfUrgences.to_excel("Urgences.xlsx", sheet_name='Urgences', index=True)
dfUrgences.dtypes
Import des données hospitalières et création d’une variable de cumul des hospitalisations « hospCumul »
Notez que le cumul est calculé comme la somme suivante : hospitalisations du jour + le nombre de retour à domicile + le nombre de décès.
Comme il y a des erreurs dans les fichiers on force la valeur du cumul de rang N à être >= à la valeur du cumul de rang N-1.
###############################################################
# Read Hospitalizations file :
# i.e donnnees-hospitalieres-covid19-YYYY-MM-DD-19h00.csv
###############################################################
dateparse = lambda x: pd.to_datetime(x, format='%d/%m/%Y')
df = pd.read_csv(hospFile, sep=";" ,
usecols= ["dep","sexe", "jour","hosp", "rea", "rad", "dc"],
dtype={"dep": str},
parse_dates= ["jour"], date_parser=dateparse)
df.dtypes
df.rename(columns={"jour": "day" }, inplace=True)
#df['dep'].astype(str) #force dep to string #done in import
df['dep'] = df['dep'].str.strip() #strip white spaces
#select global data
df = df.loc[df['sexe'] == 0]
#remove sex column
dfHospitalizations = df.drop(columns=['sexe'])
#Remove well known error dep = Na for 2020-03-24
dfHospitalizations.dropna(subset=['dep'],inplace=True)
#Sort it
dfHospitalizations.sort_values(['dep', 'day'], ascending=[True, True], inplace=True)
#Reindex
dfHospitalizations.reset_index(inplace=True, drop=True) #reset index
#hospCumul = max previous value or hosp + rad + dc
dfHospitalizations["hospCumul"]=0.0
previousDep = "00"
previousCumul=0.0
for index, row in dfHospitalizations.iterrows():
print("index:", index)
print("dep:", row['dep'])
print("date:", row['day'])
if (index==0) | (row['dep']!=previousDep):
previousCumul = dfHospitalizations.loc[index, 'hospCumul'] = row["hosp"]+row["rad"]+row["dc"]
print("First line")
else :
dfHospitalizations.loc[index, 'hospCumul'] = max(previousCumul, row["hosp"]+row["rad"]+row["dc"])
previousCumul = dfHospitalizations.loc[index, 'hospCumul']
print("other lines")
previousDep = row['dep']
print("previousCumul:", previousCumul)
#save hospitalizations if needed
#dfHospitalizations.to_excel("Hospitalizations.xlsx", sheet_name='Hospitalizations.xlsx', index=True)
dfHospitalizations.dtypes
Jonction des données d’Urgences et d’Hospitalisations
NB : on joint avec Urgences à gauche car il contient le plus de jours.
###################################################################### # Merge Urgences and Hospitalizations based on Departements and days ###################################################################### dfUrgHosp = pd.merge(dfUrgences, dfHospitalizations, on = ['dep', 'day'], how="left") dfUrgHosp.dtypes dfUrgHosp["hospCumul"] = dfUrgHosp["hospCumul"].fillna(0) #remove NaN dfUrgHosp["hosp"] = dfUrgHosp["hosp"].fillna(0) #remove NaN dfUrgHosp["rea"] = dfUrgHosp["rea"].fillna(0) #remove NaN dfUrgHosp["rad"] = dfUrgHosp["rad"].fillna(0) #remove NaN dfUrgHosp["dc"] = dfUrgHosp["dc"].fillna(0) #remove NaN
Si l’on sauvegarde les données à ce stade, on verra que les données issues du fichier des données hospitalières sont à 0 entre le 24/02/2020 et le 17/03/2020.
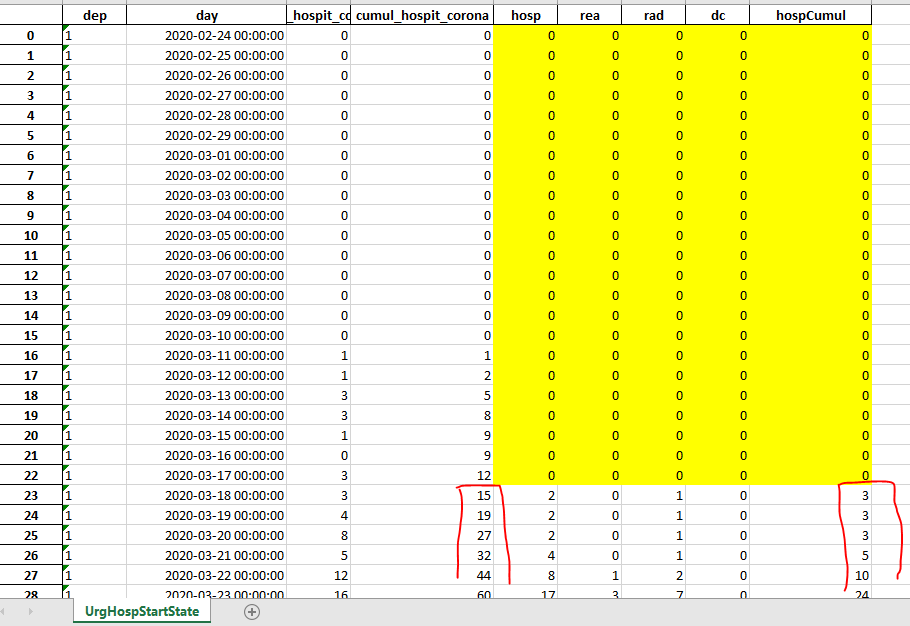
Notez aussi que les cumuls des urgences ne sont pas forcément les mêmes que ceux des hospitalisations.
Parfois ils sont plus élevés, parfois moins, on privilégiera toutefois le cumul des données hospitalières.
Calcul d’un cumul d’hospitalisations, recalculé pour les données entre le 24/02/2020 et le 17/03/2020
On procèdera en 2 temps : tout d’abord on calculera un facteur hospCumul sur cumul_hospit_corona.
Pour les données entre le 24/02/2020 et le 17/03/2020, pour lesquelles on n’a pas la valeur de hospCumul on calculera le facteur à partir de la moyenne des facteurs des dix jours suivants.
###########################################################
# Calculate hospCumul vs cumul_hospit_corona factor
###########################################################
#Resort it day descending
dfUrgHosp.sort_values(by=['dep', 'day'], ascending=[True, False], inplace=True)
#Reindex
dfUrgHosp.reset_index(inplace=True, drop=True) #reset index
#usefull function
def nonZeroYbyX(x,y):
if x>0 :
return y/x
else:
return 0
dfUrgHosp['hospCumul_cumul_hosp_corona_factor'] = 0.0
valuesForMean = []
for index, row in dfUrgHosp.iterrows():
print("index:", index)
print("dep:", row['dep'])
print("date:", row['day'])
if (index==0) | (row['dep']!=previousDep):
valuesForMean = [] #new value for mean depending on dep
dfUrgHosp.loc[index, 'hospCumul_cumul_hosp_corona_factor'] = nonZeroYbyX(row["cumul_hospit_corona"],row["hospCumul"])
print("First line")
else :
if row['day'] > datetime(2020, 3, 17, 0, 0, 0) :
dfUrgHosp.loc[index, 'hospCumul_cumul_hosp_corona_factor'] = nonZeroYbyX(row["cumul_hospit_corona"],row["hospCumul"])
else :
dfUrgHosp.loc[index, 'hospCumul_cumul_hosp_corona_factor'] = statistics.mean(valuesForMean[-10:])
valuesForMean.append(dfUrgHosp.loc[index, 'hospCumul_cumul_hosp_corona_factor'])
previousDep = row['dep']
print("valuesForMean:", valuesForMean)
NB : comme on construit le facteur à partir des données les plus récentes on inverse le tri par jour : du plus récent au plus ancien.
On crée aussi la fonction nonZeroYbyX(x,y) pour éviter les divisions par zéro si cumul_hosp_corona est nul.
Ensuite, on recrée la variable hospCumul en hospCumulRecalculé en multipliant hosp_cumul_corona par le facteur précédent.
Les valeurs après le 17/03/2020 ne changent pas.
###########################################################
# ReCalculate hospCumul from 24/02 to today hospCumulRec
###########################################################
dfUrgHosp['hospCumulRec'] = 0.0
nextHospCumulRec = 0.0
for index, row in dfUrgHosp.iterrows():
print("index:", index)
print("dep:", row['dep'])
print("date:", row['day'])
if (index==0) | (row['dep']!=previousDep) | (row['day'] > datetime(2020, 3, 17, 0, 0, 0)): #here began with the most recent day
nextHospCumulRec = 0.0
if row['hospCumul_cumul_hosp_corona_factor']>0 :
dfUrgHosp.loc[index, 'hospCumulRec'] = round(row["cumul_hospit_corona"]*row['hospCumul_cumul_hosp_corona_factor'],0)
else :
dfUrgHosp.loc[index, 'hospCumulRec'] = row['hospCumul']
print("Most recents dates")
else : #before 17/03/20
if row['hospCumul_cumul_hosp_corona_factor']>0 :
dfUrgHosp.loc[index, 'hospCumulRec'] = min(nextHospCumulRec,round(row["cumul_hospit_corona"]*row['hospCumul_cumul_hosp_corona_factor'],0))
else :
dfUrgHosp.loc[index, 'hospCumulRec'] = row['hospCumul']
nextHospCumulRec = dfUrgHosp.loc[index, 'hospCumulRec']
previousDep = row['dep']
print(" nextHospCumulRec:", nextHospCumulRec)
Recalcul d’un cumul des retours à domicile, recalculé pour avoir des données entre le 24/02/2020 et le 17/03/2020
On va faire comme précédemment en calculant un facteur rad (retours à domicile cumulés) sur hospCumulRec (les hospitalisations cumulées).
###########################################################
# rad : Retour à Domicile : Go back home
###########################################################
# Calculate Rad (Cumul) vs hospCumulRec factor
###########################################################
dfUrgHosp['rad_hospCumulRec_factor'] = 0.0
valuesForMean = []
for index, row in dfUrgHosp.iterrows():
print("index:", index)
print("dep:", row['dep'])
print("date:", row['day'])
if (index==0) | (row['dep']!=previousDep):
valuesForMean = [] #new valuez for mean depending on dep
dfUrgHosp.loc[index, 'rad_hospCumulRec_factor'] = nonZeroYbyX(row["hospCumulRec"],row["rad"])
print("First line")
else :
if row['day'] > datetime(2020, 3, 17, 0, 0, 0) :
dfUrgHosp.loc[index, 'rad_hospCumulRec_factor'] = nonZeroYbyX(row["hospCumulRec"],row["rad"])
else :
dfUrgHosp.loc[index, 'rad_hospCumulRec_factor'] = statistics.mean(valuesForMean[-10:])
valuesForMean.append(dfUrgHosp.loc[index, 'rad_hospCumulRec_factor'])
previousDep = row['dep']
print("valuesForMean:", valuesForMean)
###########################################################
# ReCalculate rad (cumul) from 24/02 to today radCumulRec
###########################################################
dfUrgHosp['radCumulRec'] = 0.0
nextRadCumulRec = 0.0
for index, row in dfUrgHosp.iterrows():
print("index:", index)
print("dep:", row['dep'])
print("date:", row['day'])
if (index==0) | (row['dep']!=previousDep) | (row['day'] > datetime(2020, 3, 17, 0, 0, 0)): #here began with the most recent day
nextRadCumulRec = 0.0
if row['rad_hospCumulRec_factor']>0 :
dfUrgHosp.loc[index, 'radCumulRec'] = round(row["hospCumulRec"]*row['rad_hospCumulRec_factor'],0)
else :
dfUrgHosp.loc[index, 'radCumulRec'] = row['rad']
print("Most recents dates")
else : #before 17/03/20
if row['rad_hospCumulRec_factor']>0 :
dfUrgHosp.loc[index, 'radCumulRec'] = min(nextRadCumulRec,round(row["hospCumulRec"]*row['rad_hospCumulRec_factor'],0))
else :
dfUrgHosp.loc[index, 'radCumulRec'] = row['rad']
nextRadCumulRec = dfUrgHosp.loc[index, 'radCumulRec']
previousDep = row['dep']
print(" nextRadCumulRec:", nextRadCumulRec)
Idem pour les décès
###########################################################
# dc : décès : Dead
###########################################################
# Calculate dc (Cumul) vs hospCumulRec factor
###########################################################
dfUrgHosp['dc_hospCumulRec_factor'] = 0.0
valuesForMean = []
for index, row in dfUrgHosp.iterrows():
print("index:", index)
print("dep:", row['dep'])
print("date:", row['day'])
if (index==0) | (row['dep']!=previousDep):
valuesForMean = [] #new valuez for mean depending on dep
dfUrgHosp.loc[index, 'dc_hospCumulRec_factor'] = nonZeroYbyX(row["hospCumulRec"],row["dc"])
print("First line")
else :
if row['day'] > datetime(2020, 3, 17, 0, 0, 0) :
dfUrgHosp.loc[index, 'dc_hospCumulRec_factor'] = nonZeroYbyX(row["hospCumulRec"],row["dc"])
else :
dfUrgHosp.loc[index, 'dc_hospCumulRec_factor'] = statistics.mean(valuesForMean[-10:])
valuesForMean.append(dfUrgHosp.loc[index, 'dc_hospCumulRec_factor'])
previousDep = row['dep']
print("valuesForMean:", valuesForMean)
###########################################################
# ReCalculate dc (cumul) from 24/02 to today dcCumulRec
###########################################################
dfUrgHosp['dcCumulRec'] = 0.0
nextdcCumulRec = 0.0
for index, row in dfUrgHosp.iterrows():
print("index:", index)
print("dep:", row['dep'])
print("date:", row['day'])
if (index==0) | (row['dep']!=previousDep) | (row['day'] > datetime(2020, 3, 17, 0, 0, 0)): #here began with the most recent day
nextdcCumulRec = 0.0
if row['dc_hospCumulRec_factor']>0 :
dfUrgHosp.loc[index, 'dcCumulRec'] = round(row["hospCumulRec"]*row['dc_hospCumulRec_factor'],0)
else :
dfUrgHosp.loc[index, 'dcCumulRec'] = row['dc']
print("Most recents dates")
else : #before 17/03/20
if row['dc_hospCumulRec_factor']>0 :
dfUrgHosp.loc[index, 'dcCumulRec'] = min(nextdcCumulRec,round(row["hospCumulRec"]*row['dc_hospCumulRec_factor'],0))
else :
dfUrgHosp.loc[index, 'dcCumulRec'] = row['dc']
nextdcCumulRec = dfUrgHosp.loc[index, 'dcCumulRec']
previousDep = row['dep']
print(" nextdcCumulRec:", nextdcCumulRec)
Recalcul des hospitalisations un jour donné à partir des hospitalisations cumulées – les retours à domicile cumulés et les décès cumulés.
Ici on utilisera un « apply ».
################################################################ # Recalculate Hosp Stock one day : hospDRec with an apply ################################################################ dfUrgHosp["hospDRec"] = dfUrgHosp.apply(lambda x: (x["hospCumulRec"] - x["radCumulRec"] - x["dcCumulRec"]), axis=1)
Recalcul du nombre de réanimations un jour donné à partir du facteur rea sur hospDrec
# reaDRec : recalculate rea (stock one day)
###########################################################
# Calculate rea (stock) vs hospDRec (stock) factor
###########################################################
dfUrgHosp['rea_hospDrec_factor'] = 0.0
valuesForMean = []
for index, row in dfUrgHosp.iterrows():
print("index:", index)
print("dep:", row['dep'])
print("date:", row['day'])
if (index==0) | (row['dep']!=previousDep):
valuesForMean = [] #new valuez for mean depending on dep
dfUrgHosp.loc[index, 'rea_hospDrec_factor'] = nonZeroYbyX(row["hospDRec"],row["rea"])
print("First line")
else :
if row['day'] > datetime(2020, 3, 17, 0, 0, 0) :
dfUrgHosp.loc[index, 'rea_hospDrec_factor'] = nonZeroYbyX(row["hospDRec"],row["rea"])
else :
dfUrgHosp.loc[index, 'rea_hospDrec_factor'] = statistics.mean(valuesForMean[-10:])
valuesForMean.append(dfUrgHosp.loc[index, 'rea_hospDrec_factor'])
previousDep = row['dep']
print("valuesForMean:", valuesForMean)
###########################################################
# ReCalculate rea (stock) from 24/02 to today reaDRec
###########################################################
dfUrgHosp['reaDRec'] = 0.0
nextReaDRec = 0.0
for index, row in dfUrgHosp.iterrows():
print("index:", index)
print("dep:", row['dep'])
print("date:", row['day'])
if (index==0) | (row['dep']!=previousDep) | (row['day'] > datetime(2020, 3, 17, 0, 0, 0)): #here began with the most recent day
nextReaDRec = 0.0
if row['rea_hospDrec_factor']>0 :
dfUrgHosp.loc[index, 'reaDRec'] = round(row["hospDRec"]*row['rea_hospDrec_factor'],0)
else :
dfUrgHosp.loc[index, 'reaDRec'] = row['rea']
print("Most recents dates")
else : #before 17/03/20
if row['rea_hospDrec_factor']>0 :
dfUrgHosp.loc[index, 'reaDRec'] = min(nextdcCumulRec,round(row["hospDRec"]*row['rea_hospDrec_factor'],0))
else :
dfUrgHosp.loc[index, 'reaDRec'] = row['rea']
nextReaDRec = dfUrgHosp.loc[index, 'reaDRec']
previousDep = row['dep']
print("nextReaDRec:", nextReaDRec)
Calcul des réanimations cumulées
Pour procéder à ce calcul, nous aurons besoin d’une étape intermédiaire pour avoir un facteur « approchant » d’un comparatif de cumuls : par exemple un facteur faisant intervenir les sommes.
##########################################################################################
# calculate hospDRecSum and reaDRecSum
##########################################################################################
# reaCumulRec is calculate using hospCumulRec * reaDRecSum_hospDRecSum_factor
dfUrgHosp.dtypes
#Resort it day ascending
dfUrgHosp.sort_values(by=['dep', 'day'], ascending=[True, True], inplace=True)
#Reindex
dfUrgHosp.reset_index(inplace=True, drop=True) #reset index
#initialize
previousDep = "00"
dfUrgHosp['hospDRecSum'] = 0.0
dfUrgHosp['reaDRecSum'] = 0.0
previousHospDRecSum = 0.0
previousReaDRecSum = 0.0
#calculate
for index, row in dfUrgHosp.iterrows():
print("index:", index)
print("dep:", row['dep'])
print("date:", row['day'])
print("hospDRec:", row['hospDRec'])
if (index==0) | (row['dep']!=previousDep):
previousHospDRecSum = 0.0
previousReaDRecSum = 0.0
dfUrgHosp.loc[index, 'hospDRecSum'] = row['hospDRec']
dfUrgHosp.loc[index, 'reaDRecSum'] = row['reaDRec']
print("First line")
else :
dfUrgHosp.loc[index, 'hospDRecSum'] = row['hospDRec'] + previousHospDRecSum
dfUrgHosp.loc[index, 'reaDRecSum'] = row['reaDRec'] + previousReaDRecSum
print("other lines")
previousDep = row['dep']
previousHospDRecSum = dfUrgHosp.loc[index, 'hospDRecSum']
previousReaDRecSum = dfUrgHosp.loc[index, 'reaDRecSum']
print(" previousreaDRecSum:", previousReaDRecSum)
##########################################################################################
# Calculate reaDRecSum_hospDRecSum_factor using an apply
##########################################################################################
#Resort it day ascending to be sure
dfUrgHosp.sort_values(by=['dep', 'day'], ascending=[True, True], inplace=True)
#Reindex
dfUrgHosp.reset_index(inplace=True, drop=True) #reset index
#calculate using an apply
dfUrgHosp['reaDRecSum_hospDRecSum_factor'] = dfUrgHosp.apply(lambda x: nonZeroYbyX(dfUrgHosp.loc[index, 'hospDRecSum'],dfUrgHosp.loc[index, 'reaDRecSum']), axis=1)
On va ensuite utiliser ce facteur pour avoir les réanimations cumulées à partir des hospitalisations cumulées.
##########################################################################################
# ReaCumulRec
##########################################################################################
# reaCumulRec is calculate using hospCumulRec * reaDRecSum_hospDRecSum_factor
dfUrgHosp.dtypes
#Resort it day ascending
dfUrgHosp.sort_values(by=['dep', 'day'], ascending=[True, True], inplace=True)
#Reindex
dfUrgHosp.reset_index(inplace=True, drop=True) #reset index
#uinitialize
previousDep = "00"
dfUrgHosp['reaCumulRec'] = 0.0
previousReaCumulRec = 0.0
#calculate .
for index, row in dfUrgHosp.iterrows():
print("index:", index)
print("dep:", row['dep'])
print("date:", row['day'])
if (index==0) | (row['dep']!=previousDep):
previousReaCumulRec = 0.0
dfUrgHosp.loc[index, 'reaCumulRec'] = round(row["hospCumulRec"]*dfUrgHosp.loc[index, 'reaDRecSum_hospDRecSum_factor'],0)
print("First line")
else :
dfUrgHosp.loc[index, 'reaCumulRec'] = max(previousReaCumulRec , round(row["hospCumulRec"]*dfUrgHosp.loc[index, 'reaDRecSum_hospDRecSum_factor'],0))
print("other lines")
previousDep = row['dep']
previousReaCumulRec = dfUrgHosp.loc[index, 'reaCumulRec']
print(" previousReaCumulRec:", previousReaCumulRec)
Calcul des données d’incidences
Au final nous allons calculer les données d’incidences newHosp, newRea, newReaOut, newRad et newDc à partir de données de cumuls ou de « stocks » (nombre un jour donné).
###################################################################################################
# calculate newHosp, newRea, newReaOut, newRad, newDc from max0,Current Cumuls - previous Cumuls)
# except for newReaOut = max(0, Previous ReaDRec - current ReaDrec)
###################################################################################################
#initialize columns
dfUrgHosp['newHosp'] = 0.0
dfUrgHosp['newRea'] = 0.0
dfUrgHosp['newReaOut'] = 0.0
dfUrgHosp['newRad'] = 0.0
dfUrgHosp['newDc'] = 0.0
#initaize previous variables:
previousDep = "00"
previousHospCumulRec = 0.0
previousReaCumulRec = 0.0
previousReaDRec = 0.0 #for newReaOut
previousRadCumulRec = 0.0
previousDcCumulRec = 0.0
#calculate
for index, row in dfUrgHosp.iterrows():
#print("index:", index)
#print("dep:", row['dep'])
#print("date:", row['day'])
if (index==0) | (row['dep']!=previousDep):
dfUrgHosp.loc[index, 'newHosp'] = row['hospCumulRec']
dfUrgHosp.loc[index, 'newRea'] = row['reaCumulRec']
dfUrgHosp.loc[index, 'newReaOut'] = row['reaDRec']
dfUrgHosp.loc[index, 'newRad'] = row['radCumulRec']
dfUrgHosp.loc[index, 'newDc'] = row['dcCumulRec']
print("First line newDc :", dfUrgHosp.loc[index, 'newDc'])
else :
dfUrgHosp.loc[index, 'newHosp'] = max(0,row['hospCumulRec'] - previousHospCumulRec)
dfUrgHosp.loc[index, 'newRea'] = max(0,row['reaCumulRec'] - previousReaCumulRec)
dfUrgHosp.loc[index, 'newReaOut'] = max(0,previousReaDRec - row['reaDRec'])
dfUrgHosp.loc[index, 'newRad'] = max(0,row['radCumulRec'] - previousRadCumulRec)
dfUrgHosp.loc[index, 'newDc'] = max(0,row['dcCumulRec'] - previousDcCumulRec)
#print("previousDcCumulRec :", previousDcCumulRec)
print("Other Lines newDc :", dfUrgHosp.loc[index, 'newDc'])
previousHospCumulRec = row['hospCumulRec']
previousReaCumulRec = row['reaCumulRec']
previousReaDRec = row['reaDRec']
previousRadCumulRec = row['radCumulRec']
previousDcCumulRec = row['dcCumulRec']
previousDep = row['dep']
#####################################################################
# Save in Excel to check, if needed
#####################################################################
#Resort it day ascending
dfUrgHosp.sort_values(by=['dep', 'day'], ascending=[True, True], inplace=True)
#Reindex
dfUrgHosp.reset_index(inplace=True, drop=True) #reset index
dfUrgHosp.to_excel("dfUrgHosp.xlsx", sheet_name='UrgHosp', index=True)
Au final on obtient le fichier suivant (vu précédemment)

Les données souhaitées démarrent bien à partir du 24/02/2020
Partie II : calcul du nombre de jours en réanimation par département
C’est dans cette partie que nous allons ranger les données en un jeu exploitable avec une ligne par hospitalisation et les dates en colonnes.
Comme nous l’indiquions précédemment, nous allons pour cela utiliser le hasard, ce qui va nous obliger à créer plusieurs échantillons pour dégager une moyenne cohérente.
On peut par exemple créer 100 échantillons (ce qui est un peu long), 20 donnent déjà des résultats intéressants.
Le choix dépendra de la puissance de votre machine. Sinon vous pouvez aussi utiliser Google Colab (je n’ai pas essayé).
Le jeu de données qui contiendra tous les échantillons rangés est nommé dfTidyHosp. Quand c’est le cas on rajoutera le nombre de jour en réanimation pour cette hospitalisation.
Des moyennes par échantillons seront calculées pour chaque échantillon dans les jeux de données dfDepDaysInRea.
A partir de ces échantillons rangés, on calculera des nombre de jours de réanimations pour chaque échantillon et chaque département. Il s’agit du jeux de données dfAllDepDaysInRea
les jeux dfTidyHospXX, dfDepDaysInReaXX et dfAllDepDaysInRea sont calculés dans la même boucle de programme.
Au final on calculera une moyenne pondérée du nombre de jours de réanimations par département, sur l’ensemble des échantillons : dfDaysInReaByDep
initialisation et début de la boucle
# II - calculate number of days in Rea
##################################################################################
##############################################################################
# Read file with newHosp, newRea , newReaOut, newRad, newDc data
##############################################################################
dfNewHosp = pd.read_excel("dfUrgHosp.xlsx", sheet_name='UrgHosp', usecols=["dep", "day", "newHosp", "newRea", "newReaOut", "newDc", "newRad"])
dfNewHosp.dtypes
maxSamples = 100 #number of samples 100 is very good, 20 is enough
column_names = ["numSample", "dep", "dayInReaNZMean", "countDayInReaNZ", "DaysInReaSampleWMean"]
dfAllDepDaysInRea = pd.DataFrame(columns = column_names)
for numSample in range(0,maxSamples) :
print("numSample:", numSample)
#Create a Tidy Data Data Frame : one row for each hospitalization
column_names = ["dep", "dayHosp", "dayRea", "dayReaOut", "dayDc", "dayRad", "closed"]
dfTidyHosp = pd.DataFrame(columns = column_names)
#dfTidyHosp.dtypes
#Normally sorted but force it
dfNewHosp.sort_values(['dep', 'day'], ascending=[True, True], inplace=True)
#dfNewHosp.dtypes
Création des lignes d’hospitalisations en fonction du nombre de nouvelles hospitalisations ce jour.
#First Create the Tidy rows from new hospitalizations
#Add new rows for new days
for index, row in dfNewHosp.iterrows():
#print("index:",index)
#add new hospitalizations
if row['newHosp']>0:
#print("dep", row["dep"])
#print("jour", row["day"])
dfOneTidyHosp = pd.DataFrame({"dep" : [row["dep"]],
"dayHosp" : [row["day"]],
"dayRea" : datetime(1970, 1, 1, 0, 0, 0),
"dayReaOut" : datetime(1970, 1, 1, 0, 0, 0),
"dayDc" : datetime(1970, 1, 1, 0, 0, 0),
"dayRad" : datetime(1970, 1, 1, 0, 0, 0),
"closed": False}) #New work Tidy
#☺Splits Rows
if row['newHosp']>1:
dfOneTidyHosp = dfOneTidyHosp.loc[dfOneTidyHosp.index.repeat(row['newHosp'])]
dfOneTidyHosp.reset_index(inplace=True, drop=True) #reset index
dfTidyHosp = pd.concat([dfTidyHosp,dfOneTidyHosp]) #new rows
#reset index for global Tidy hosp dataframe
dfTidyHosp.reset_index(inplace=True, drop=True) #reset index
Ajout des dates de réanimations dans les hospitalisations au hasard
#############################################################
#Add newRea
if row['newRea']>0:
#select not already in Rea
for i in range(0,int(row['newRea'])) :
#print("i rea:",i)
#select not in rea
dfTidyIndex = dfTidyHosp.loc [ (dfTidyHosp["dep"] == row["dep"]) &
(dfTidyHosp["dayRea"] == datetime(1970, 1, 1, 0, 0, 0)) &
(dfTidyHosp["dayReaOut"] == datetime(1970, 1, 1, 0, 0, 0)) &
(dfTidyHosp["dayDc"] == datetime(1970, 1, 1, 0, 0, 0)) &
(dfTidyHosp["dayRad"] == datetime(1970, 1, 1, 0, 0, 0)) &
(dfTidyHosp["closed"] == False) ]
if dfTidyIndex.shape[0]>0 :
myIndex = random.randint(0,dfTidyIndex.shape[0]-1)
print("dfTidyIndex0Rea:",dfTidyIndex.index[myIndex])
dfTidyHosp["dayRea"].iloc[dfTidyIndex.index[ myIndex]]= row["day"]
break
Ajout des dates de sorties de réanimations au hasard dans les réanimations
#####################################
#Add newReaOut
if row['newReaOut']>0:
#print("lits de suite:",row['newReaOut'])
#print("dep", row["dep"])
#print("jour", row["day"])
#select only in Rea
for i in range(0,int(row['newReaOut'])) :
#print("i lits de suite:",i)
#select only in rea
dfTidyIndex = dfTidyHosp.loc [ (dfTidyHosp["dep"] == row["dep"]) &
(dfTidyHosp["dayRea"] != datetime(1970, 1, 1, 0, 0, 0)) &
(dfTidyHosp["dayReaOut"] == datetime(1970, 1, 1, 0, 0, 0)) &
(dfTidyHosp["dayDc"] == datetime(1970, 1, 1, 0, 0, 0)) &
(dfTidyHosp["dayRad"] == datetime(1970, 1, 1, 0, 0, 0)) &
(dfTidyHosp["closed"] == False) ]
if dfTidyIndex.shape[0]>0 :
myIndex = random.randint(0,dfTidyIndex.shape[0]-1)
print("dfTidyIndex0ReaOut:",dfTidyIndex.index[myIndex])
dfTidyHosp["dayReaOut"].iloc[dfTidyIndex.index[myIndex]]= row["day"]
break
Ajout des dates de retour à domicile « au hasard »
Ce n’est pas tout à fait vrai car on va privilégier en premier les personnes qui ne sont pas passées en réanimation.
#Add newRad two choices
if row['newRad']>0:
#select first no Rea !!! Proba is more certain
for i in range(0,int(row['newRad'])) :
#print("i rad:",i)
#select first no rea
dfTidyIndex = dfTidyHosp.loc [ (dfTidyHosp["dep"] == row["dep"]) &
(dfTidyHosp["dayRea"] == datetime(1970, 1, 1, 0, 0, 0)) &
(dfTidyHosp["dayDc"] == datetime(1970, 1, 1, 0, 0, 0)) &
(dfTidyHosp["dayRad"] == datetime(1970, 1, 1, 0, 0, 0)) &
(dfTidyHosp["closed"] == False) ]
if dfTidyIndex.shape[0]>0 :
myIndex = random.randint(0,dfTidyIndex.shape[0]-1)
#print("myIndex:", myIndex)
print("dfTidyIndex0RadNoRea:",dfTidyIndex.index[myIndex])
dfTidyHosp["dayRad"].iloc[dfTidyIndex.index[myIndex]]= row["day"]
dfTidyHosp["closed"].iloc[dfTidyIndex.index[myIndex]]= True
break
#next select Rea or not but <= row["day"]
for j in range(i,int(row['newRad'])) :
#print("i rad rea or not:",i)
#select Rea <= row["day"]
dfTidyIndex = dfTidyHosp.loc [ (dfTidyHosp["dep"] == row["dep"]) &
(dfTidyHosp["dayRea"] <= row["day"] ) &
(dfTidyHosp["dayReaOut"] <= row["day"] ) &
(dfTidyHosp["dayDc"] == datetime(1970, 1, 1, 0, 0, 0)) &
(dfTidyHosp["dayRad"] == datetime(1970, 1, 1, 0, 0, 0)) &
(dfTidyHosp["closed"] == False) ]
if dfTidyIndex.shape[0]>0 :
myIndex = random.randint(0,dfTidyIndex.shape[0]-1)
#print("myIndex:", myIndex)
print("dfTidyIndex0RadRea:",dfTidyIndex.index[myIndex])
dfTidyHosp["dayRad"].iloc[dfTidyIndex.index[myIndex]]= row["day"]
dfTidyHosp["closed"].iloc[dfTidyIndex.index[myIndex]]= True
break
Ajout des dates de décès « au hasard »
Le « hasard » n’est pas tout à fait vrai non plus, mais cette fois on va « privilégier » les personnes qui sont passées en réanimation en premier.
#Add newDc
if row['newDc']>0:
#select first Rea only #proba is up to die
for i in range(0,int(row['newDc'])) :
#print("i Dc:",i)
#select first Rea
dfTidyIndex = dfTidyHosp.loc [ (dfTidyHosp["dep"] == row["dep"]) &
(dfTidyHosp["dayRea"] > datetime(1970, 1, 1, 0, 0, 0)) &
(dfTidyHosp["dayDc"] == datetime(1970, 1, 1, 0, 0, 0)) &
(dfTidyHosp["dayRad"] == datetime(1970, 1, 1, 0, 0, 0)) &
(dfTidyHosp["closed"] == False) ]
if dfTidyIndex.shape[0]>0 :
myIndex = random.randint(0,dfTidyIndex.shape[0]-1)
print("dfTidyIndex0DcRea:",dfTidyIndex.index[myIndex])
dfTidyHosp["dayDc"].iloc[dfTidyIndex.index[myIndex]]= row["day"]
dfTidyHosp["closed"].iloc[dfTidyIndex.index[myIndex]]= True
break
#next select Rea or not
for j in range(i,int(row['newDc'])) :
#print("i rea:",i)
#select not Rea or Not
dfTidyIndex = dfTidyHosp.loc [ (dfTidyHosp["dep"] == row["dep"]) &
(dfTidyHosp["dayDc"] == datetime(1970, 1, 1, 0, 0, 0)) &
(dfTidyHosp["dayRad"] == datetime(1970, 1, 1, 0, 0, 0)) &
(dfTidyHosp["closed"] == False) ]
if dfTidyIndex.shape[0]>0 :
myIndex = random.randint(0,dfTidyIndex.shape[0]-1)
print("dfTidyIndex0DcAll:",dfTidyIndex.index[myIndex])
dfTidyHosp["dayDc"].iloc[dfTidyIndex.index[myIndex]]= row["day"]
dfTidyHosp["closed"].iloc[dfTidyIndex.index[myIndex]]= True
break
#dfTidyHosp.dtypes
Calcul du nombre de jours en réanimation pour chaque hospitalisation concernée.
#Calculate Days in Rea for this sample
def calculateDaysInRea(x,y,z,t):
#x = "dayRea"
#y = "dayReaOut"
#z = "dayDc"
#t = "dayRad"
#if rea
if x>datetime(1970, 1, 1, 0, 0, 0) :
#Rea Out ????
if y>datetime(1970, 1, 1, 0, 0, 0) :
#Rea Out OK
minOut=y
else :
#Dead ???
if z>datetime(1970, 1, 1, 0, 0, 0) :
minOut=z
else :
#Rad/ hosp out ????
if t>datetime(1970, 1, 1, 0, 0, 0) :
minOut=z
else :#still in hosp -> out is today for the moment
minOut= datetime.today()
if minOut > datetime(1970, 1, 1, 0, 0, 0) : #not sure it is necessary
myDelta = minOut-x
print("days:",myDelta.days+1 )
return myDelta.days+1
else : return None
dfTidyHosp["daysInRea"] = dfTidyHosp.apply(lambda x: calculateDaysInRea(x["dayRea"], x["dayReaOut"], x["dayDc"], x["dayRad"] ), axis=1)
strNumSample = str(numSample)
dfTidyHosp.reset_index(inplace=True, drop=True) #reset index
#Save the Sample - long to save could be in comments
print("Save the Sample Tidy")
dfTidyHosp.to_excel("TidyHosp"+str(numSample)+".xlsx", sheet_name='Tidy', index=True)
########
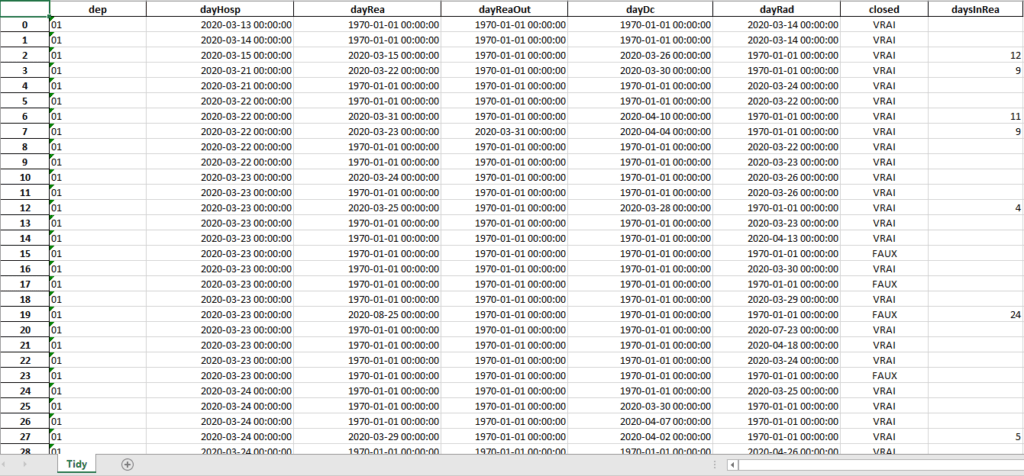
Exemple de fichier rangé TidyHosp0.xlsx :
Calcul du nombre moyen de jours en réanimation par département pour cet échantillon
#################################################################################
# Calculate number of days in rea by department for this sample
###### create meanRea DF (by Department)
uniqueDep = dfTidyHosp["dep"].unique() #
column_names = ["numSample", "dep", "dayInReaNZMean", "countDayInReaNZ", "DaysInReaSampleWMean"]
dfDepDaysInRea = pd.DataFrame(columns = column_names)
for dep in uniqueDep :
print("dep:",dep)
dfTidyHospDep = dfTidyHosp.loc [ dfTidyHosp["dep"] == dep ]
dayInReaNZMean = dfTidyHospDep["daysInRea"].mean( skipna = True)
countDayInReaNZ = dfTidyHospDep["daysInRea"].dropna().size #weight
print("dayInReaNZMean:",dayInReaNZMean)
print("countDayInReaNZ:",countDayInReaNZ)
dfOneDepDaysInRea = pd.DataFrame({"numSample": numSample, "dep" : dep, "dayInReaNZMean" : dayInReaNZMean, "countDayInReaNZ" : countDayInReaNZ}, index=[0]) #New work Tidy
dfDepDaysInRea = pd.concat([dfDepDaysInRea,dfOneDepDaysInRea]) #new rows
#calculate weighted average
dfDepDaysInRea["DaysInReaSampleWMean"] = np.average(a=dfDepDaysInRea["dayInReaNZMean"],weights=dfDepDaysInRea["countDayInReaNZ"] )
dfDepDaysInRea.reset_index(inplace=True, drop=True)
print("Save one Sample Days in Rea by Day")
#Save one sample file if needed
dfDepDaysInRea.to_excel("dfDepDaysInRea"+str(numSample)+".xlsx", sheet_name='Means', index=True)
#Concat data in all samples data
dfAllDepDaysInRea = pd.concat([dfAllDepDaysInRea,dfDepDaysInRea]) #new rows
Exemple de fichier de moyennes pour un échantillon dfDepDaysInRea0.xlsx :
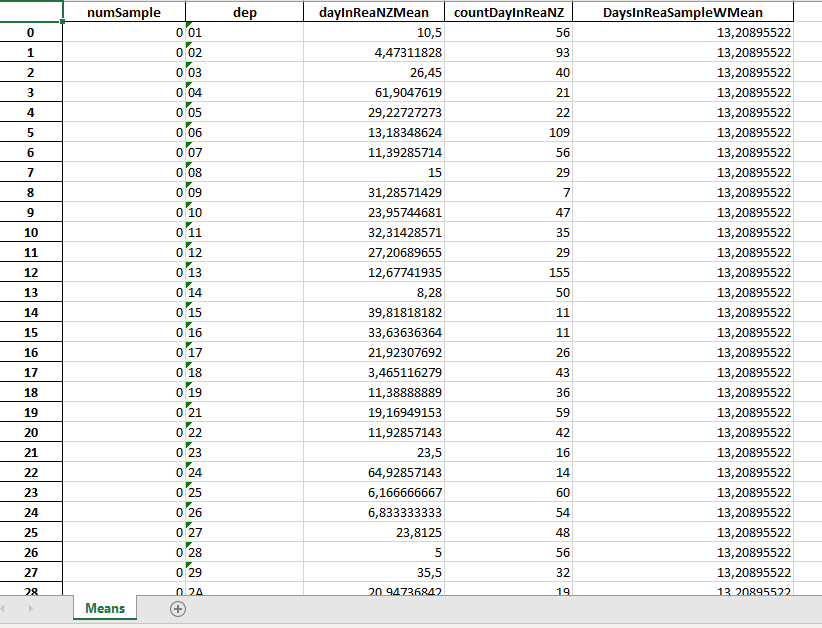
DaysinReaSampleWmean est la moyenne pondérée par le nombre de cas de réanimations par département (counDayInReaNZ) pour la France. Par exemple ici la moyenne a été d’environ 13 jours en réanimation en France
Sauvegarde des échantillons dans un seul fichier
print("Save all Samples Days in Rea by Day")
#Calculate Global average with weignt
dfAllDepDaysInRea["DaysInReaGlobalWMean"] = np.average(a=dfAllDepDaysInRea["dayInReaNZMean"],weights=dfAllDepDaysInRea["countDayInReaNZ"] )
dfAllDepDaysInRea.reset_index(inplace=True, drop=True)
dfAllDepDaysInRea.to_excel("dfAllDepDaysInRea.xlsx", sheet_name='Means', index=True)
Le fichier de sauvegarde de tous les échantillons à le format suivant :
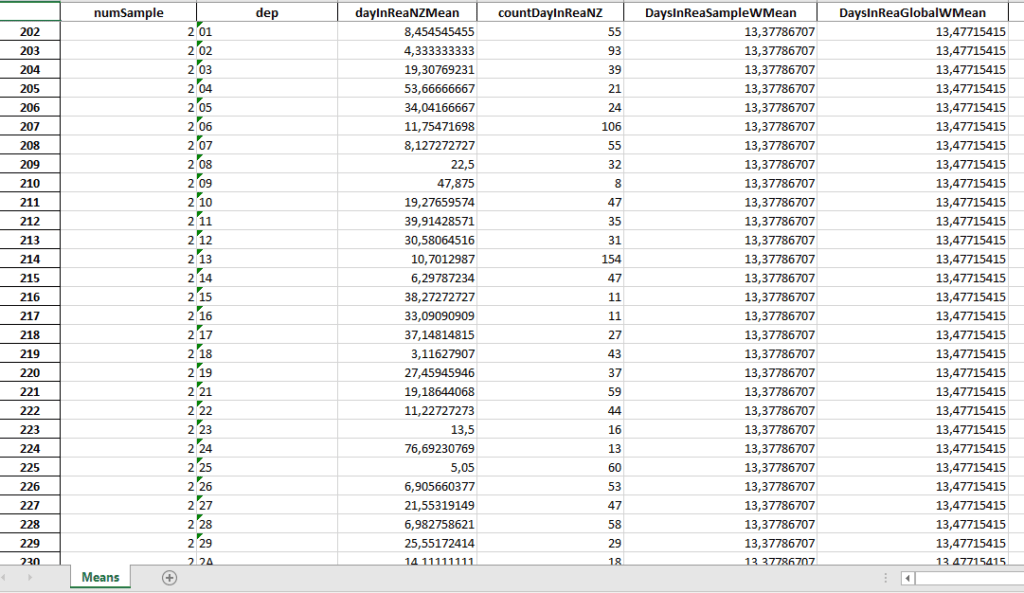
On a rajouté une moyenne globale pondérée de tous les échantillons pour la France, DayInReaGlobaWMean, qui vaut ici 13,47 jours
Création du jeu de données / fichier final des moyennes pondérées de tous les échantillons par département.
####################################################################################################
# FINAL FILE
# Global mean of samples by dep
####################################################################################################
#Resort by dep
dfAllDepDaysInRea.dtypes
dfAllDepDaysInRea['dep']= dfAllDepDaysInRea['dep'].astype(str) #force dep to string
dfAllDepDaysInRea['dep'] = dfAllDepDaysInRea['dep'].str.strip() #strip white spaces
dfAllDepDaysInRea.sort_values(by='dep', ascending=True, inplace=True)
#reindex
dfAllDepDaysInRea.reset_index(inplace=True, drop=True)
#createnew df for days in Rea by dep for all samples
column_names = ["dep", "daysInReaByDep"]
dfDaysInReaByDep = pd.DataFrame(columns = column_names)
uniqueDep = dfAllDepDaysInRea["dep"].unique() #
for dep in uniqueDep :
print("dep:",dep)
#create one dataframe for one dep
daysInReaByDep = 0.0 #initialize value
dfOneDaysInReaByDep = pd.DataFrame(columns = column_names)
#select data for the current dep
dfOneAllDepDaysInRea = dfAllDepDaysInRea.loc [ dfAllDepDaysInRea["dep"] == dep ]
#calculate average for this dep
daysInReaByDep = np.average(a=dfOneAllDepDaysInRea["dayInReaNZMean"],weights=dfOneAllDepDaysInRea["countDayInReaNZ"] )
dfOneDaysInReaByDep = pd.DataFrame({"dep": dep, "daysInReaByDep" : daysInReaByDep}, index=[0]) #New work df
print("average:",dfOneDaysInReaByDep["daysInReaByDep"] )
#concat in all dep dataframe
dfDaysInReaByDep = pd.concat([dfDaysInReaByDep,dfOneDaysInReaByDep]) #new rows
dfDaysInReaByDep.reset_index(inplace=True, drop=True)
dfDaysInReaByDep.to_excel("dfDaysInReaByDep.xlsx", sheet_name='Days', index=False)
Le fichier obtenu a le format suivant :
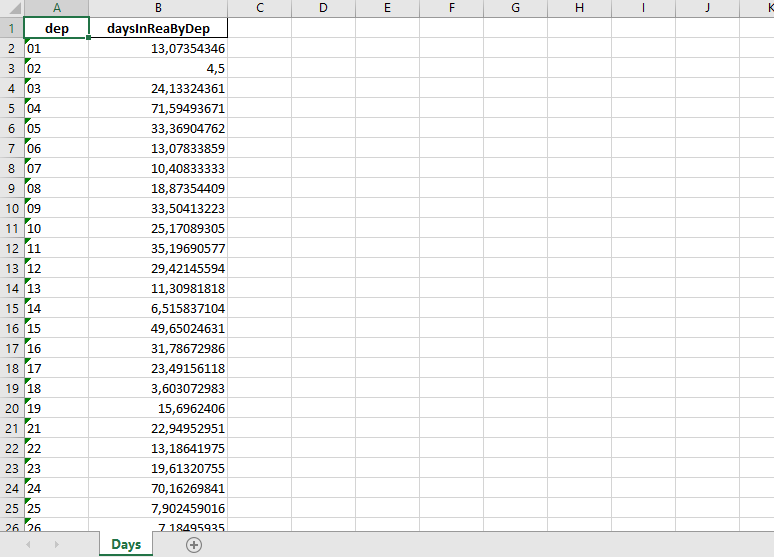
Ce fichier pourrait être utilisé par la suite dans une nouvelle étude comparative du taux de mortalité du Covid19 par département.
A bientôt,
Commentaires, remarques et suggestions bienvenus.
Pierre