Partager la publication "Quelle est la durée de vie de mes articles sur mon site ? Python"
Cet article reprend l’essentiel des 2 articles précédents :
- Quelle est la durée de vie de mes articles sur mon site ? R – I
- Quelle est la durée de vie de mes articles sur mon site ? R – II
cette fois avec Python.
Rappelons la question que l’on, se pose : Quelle est la durée où, en moyenne, mes articles « apportent » du trafic sur mon site Web ?
Nous ne détaillerons pas toutes les explications ici, merci de vous reporter aux articles avec R au besoin.
De quoi aurons nous besoin ?
Python Anaconda
Anaconda est une version de Python dédiée aux Data Sciences. Rendez vous sur la page de téléchargement d’Anaconda Distribution pour télécharger la version qui vous convient selon votre ordinateur.
Jeu de données
Vous pouvez soit utiliser nos données pour tester le programme :
Soit, créer votre propre jeu de données à partir de vos statistiques Google Analytics, en suivant la procédure sur les articles précédents :
- Importer les données de Google Analytics API avec Python Anaconda.
- Nettoyage du Spam dans Google Analytics avec Python.
- Détection du trafic Web significatif avec Python.
- Publication d’articles vs événements significatifs en Python.
Code source :
Copiez/Collez les codes sources suivants ou récupérez gratuitement le code en entier dans notre boutique : https://www.anakeyn.com/boutique/produit/script-python-duree-de-vie-des-articles/ .
Chargement des bibliothèques utiles
Et aussi, paramétrage de votre environnement.
# -*- coding: utf-8 -*- """ Created on Fri May 31 10:50:38 2019 @author: Pierre """ ######################################################################### # ArticleLifetimePython # Durée de vie des articles sur son site # Auteur : Pierre Rouarch 2019 - Licence GPL 3 # Données : Issues de l'API de Google Analytics - # Comme illustration Nous allons travailler sur les données du site # https://www.networking-morbihan.com ############################################################# # On démarre ici pour récupérer les bibliothèques utiles !! ############################################################# #def main(): #on ne va pas utiliser le main car on reste dans Spyder #Chargement des bibliothèques utiles (décommebter au besoin) import numpy as np #pour les vecteurs et tableaux notamment import matplotlib.pyplot as plt #pour les graphiques import scipy as sp #pour l'analyse statistique import pandas as pd #pour les Dataframes ou tableaux de données import seaborn as sns #graphiques étendues import math #notamment pour sqrt() from datetime import timedelta from scipy import stats #pip install scikit-misc #pas d'install conda ??? #from skmisc import loess #pour methode Loess compatible avec stat_smooth #conda install -c conda-forge plotnine #from plotnine import * #pour ggplot like #conda install -c conda-forge mizani #from mizani.breaks import date_breaks #pour personnaliser les dates affichées #Si besoin Changement du répertoire par défaut pour mettre les fichiers de sauvegarde #dans le même répertoire que le script. import os print(os.getcwd()) #verif #mon répertoire sur ma machine - nécessaire quand on fait tourner le programme #par morceaux dans Spyder. #myPath = "C:/Users/Pierre/CHEMIN" #os.chdir(myPath) #modification du path #print(os.getcwd()) #verif
Récupération des fichiers de pages vues et des articles.
Attention ces fichiers doivent être dans le répertoire courant de votre code source.
###############################################################################
#Récupération du fichier de données des pages vues
###############################################################################
myDateToParse = ['date'] #pour parser la variable date en datetime sinon object
dfPageViews = pd.read_csv("dfPageViews.csv", sep=";", dtype={'Année':object}, parse_dates=myDateToParse)
#verifs
dfPageViews.dtypes
dfPageViews.count() #72821 enregistrements
dfPageViews.head(20)
###############################################################################
#Récupération du fichier de données articles
###############################################################################
myArticles = pd.read_csv("myArticles.csv", sep=";", dtype={'Année':object}, parse_dates=myDateToParse)
#verifs
myArticles.dtypes
myArticles.count() #95 enregistrements
myArticles.head(20)
Calcul du trafic « Articles Marketing »
Il s’agit du trafic en rapport avec les pages d’articles que l’on souhaite investiguer. On les obtient à partir du trafic des « pages de base ».
##########################################################################
# Calcul du trafic hors "articles marketing" ie "trafic de base"
# On va supprimer toutes les pages vues correspondantes aux articles
# "Marketing" ainsi que toutes les pages vues dont l'entrée s'est faite
# par un article "Marketing". on compare ensuite au traffic global.
##########################################################################
#on va créer une colonne "origin_index" qui va servir par la suite
dfPageViews['origin_index'] = np.arange(len(dfPageViews))
myArticles['pagePath']=myArticles['links'].str.replace('https://www.networking-morbihan.com', '')
pattern = '|'.join(myArticles['pagePath'])
#on enlève les pagePath
indexPagePathToKeep = dfPageViews[(dfPageViews.pagePath.str.contains(pat=pattern,regex=True)==False)].index
dfBasePageViews = dfPageViews.iloc[indexPagePathToKeep]
dfBasePageViews.reset_index(inplace=True, drop=True) #on reindexe
dfBasePageViews.count() #43633
#puis on enlève les landingPagePath
indexLandingPagePathToKeep = dfBasePageViews[(dfBasePageViews.landingPagePath.str.contains(pat=pattern,regex=True)==False)].index
dfBasePageViews = dfBasePageViews.iloc[indexLandingPagePathToKeep]
dfBasePageViews.reset_index(inplace=True, drop=True) #on reindexe
dfBasePageViews.count() #37614 idem que dans R
###############################################################################
# Sauvegardes en csv
dfBasePageViews.to_csv("dfBasePageViews.csv", sep=";", index=False) #séparateur ;
##################################################################
###########################################################################
# Calcul du trafic "articles marketing" par "anti join" du "trafic de base"
#les pages avec nos articles sont les autres
#############################################################################
dfAMPageViews = dfPageViews.drop(dfPageViews.merge(dfBasePageViews).origin_index)
dfAMPageViews.count() #35207 observations idem que dans R
len(dfAMPageViews.index)
###############################################################################
# Sauvegarde en csv
dfAMPageViews.to_csv("dfAMPageViews.csv", sep=";", index=False) #séparateur ;
###############################################################################
Distribution du trafic des articles marketing à x mois
On va créer une fonction pour déterminer cela pour n’importe quel mois.
############################################################################
# Significativité du trafic « articles marketing » dans les mois suivants
# la publication.
# il s'agit des pages visitées suite à une entrée sur une page
# "Marketing" OU une page "Marketing" elle même : AM
############################################################################
############################################################################
# Fonction pour récupérer les distributions sur un numéro de période et
# un nombre de jour
############################################################################
def getMyDistribution(myPageViews, myArticles, myNumPeriode, myLastDate, myNbrOfDays=30, myTestType="AM"):
'''
En ENTREE :
myPageViews = une dataframe de Pages vues à tester avec les variables au minimum :
-date : date YYYY-MM-DD - date de la visite sur la page"
-landingPagePath : chr path de la page d'entrée sur le site ex '/rentree-2011'"
-PagePath : chr path de la page visitée sur le site site ex '/rentree-2011'"
myArticles = une dataframe de Pages vues que l'on souhaite investiguer et qui sont parmi les précédentes avec les variables au minimum
-date : date YYYY-MM-DD - date de la visite sur la page
-PagePath : chr - path de la page visitée sur le site site ex '/rentree-2011'"
myNumPeriode : integer Numéro de période par exemple 1 si c'est la première période
myNbrOfDays : int - nombre de jours pour la période 30 par défaut
myLastDate : date YYYY-MM-DD - date limite à investiguer.
myTestType='AM' : chr - 'AM' test du landingPagePath ou pagePath sinon test du pagePath seul.
EN SORTIE
ThisPeriodPV : np.array des pages vues pour chaque page pour la période interrogée.'''
#ThisPeriodPV = np.empty([len(myArticles.index),1]) #np.array pour sauvegarder la distribution #
#ThisPeriodPV = np.empty([len(myArticles.index)])
#90
#pd.DataFrame(columns= ['ThisPeriodPV'], index=range(len(myArticles.index)), dtype='float')
dfThisPeriodPV = pd.DataFrame(columns= ['ThisPeriodPV'], index=range(len(myArticles.index)), dtype='float')
for i in range(0,len(myArticles.index)-1) :
Link = myArticles.iloc[i]["pagePath"] #lien i /
Date1 = myArticles.iloc[i]["date"]+ timedelta(days=(myNumPeriode-1)*myNbrOfDays)
Date2 = Date1+timedelta(days=myNbrOfDays)
if (myTestType == "AM") :
myPV = myPageViews.loc[(myPageViews.pagePath == Link) | (myPageViews.landingPagePath == Link)]
else :
myPV = myPageViews.loc[(myPageViews.pagePath == Link)]
#Marketing
myPVPeriode = myPV.loc[(myPV['date'] >= Date1) & (myPV['date'] <= Date2)]
myPVPeriodeLength = len(myPVPeriode)
if Date1 > myLastDate :
break #on arrête
#myPVPeriodeLength = np.nan #pour éviter d'avoir des 0 nous avions oublié cet aspect là précédemment
# dfThisPeriodPV = dfThisPeriodPV.append({'ThisPeriodPV':myPVPeriodeLength}, ignore_index=True)
dfThisPeriodPV.loc[i, 'ThisPeriodPV'] = myPVPeriodeLength
return dfThisPeriodPV #ThisPeriodPV,
#/getMyDistribution
#help(getMyDistribution) #test du help
POUR LE MOIS I :
############################################################################
# Pour tous les mois
############################################################################
lastDate = dfPageViews.iloc[len(dfPageViews.index)-1]['date'] #dernière date dans les pages vues
############################################################################
# Pour le mois 1
############################################################################
myMonthNumber = 1
dfAMThisMonthPV = getMyDistribution(myPageViews=dfAMPageViews,
myArticles=myArticles,
myNumPeriode=myMonthNumber,
myNbrOfDays=30,
myLastDate=lastDate,
myTestType="AM")
#test de normalité shapiro wilk
dfAMThisMonthPVDropNa = dfAMThisMonthPV.dropna() #
myW, myPValeur = stats.shapiro(dfAMThisMonthPVDropNa['ThisPeriodPV'].values)
myPValeur #0.0007478381157852709 normalité rejeté
sns.set() #paramètres esthétiques ressemble à ggplot par défaut.
fig, ax = plt.subplots() #un seul plot
sns.distplot(dfAMThisMonthPVDropNa['ThisPeriodPV'].values, bins=30, kde=False, rug=True)
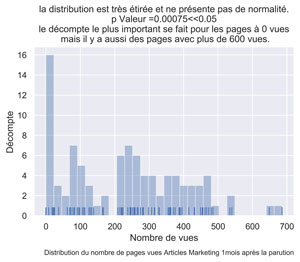
ax.set(xlabel="Nombre de vues", ylabel='Décompte',
title="la distribution est très étirée et ne présente pas de normalité.\n p Valeur =" + str(round(myPValeur,5)) +
"<<0.05 \n le décompte le plus important se fait pour les pages à 0 vues \n mais il y a aussi des pages avec plus de 600 vues.")
fig.text(.9,-.05,"Distribution du nombre de pages vues Articles Marketing " + str(myMonthNumber) + "mois après la parution",
fontsize=9, ha="right")
#plt.show()
fig.savefig("Dist-PV-AM-Mois-"+str(myMonthNumber)+".png", bbox_inches="tight", dpi=600)
POUR LE MOIS 2 :
############################################################################
# Pour le mois 2
############################################################################
myMonthNumber = 2
dfAMThisMonthPV = getMyDistribution(myPageViews=dfAMPageViews,
myArticles=myArticles,
myNumPeriode=myMonthNumber,
myNbrOfDays=30,
myLastDate=lastDate,
myTestType="AM")
#test de normalité shapiro wilk
dfAMThisMonthPVDropNa = dfAMThisMonthPV.dropna() #
myW, myPValeur = stats.shapiro(dfAMThisMonthPVDropNa['ThisPeriodPV'].values)
myPValeur #normalité rejeté
sns.set() #paramètres esthétiques ressemble à ggplot par défaut.
fig, ax = plt.subplots() #un seul plot
sns.distplot(dfAMThisMonthPVDropNa['ThisPeriodPV'].values, bins=30, kde=False, rug=True)
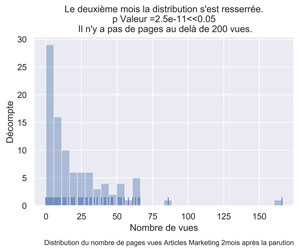
ax.set(xlabel="Nombre de vues", ylabel='Décompte',
title="Le deuxième mois la distribution s'est resserrée.\n p Valeur =" + str(round(myPValeur,12)) +
"<<0.05 \n Il n'y a pas de pages au delà de 200 vues.")
fig.text(.9,-.05,"Distribution du nombre de pages vues Articles Marketing " + str(myMonthNumber) + "mois après la parution",
fontsize=9, ha="right")
#plt.show()
fig.savefig("Dist-PV-AM-Mois-"+str(myMonthNumber)+".png", bbox_inches="tight", dpi=600)
POUR LE MOIS 10 :
############################################################################
# Pour le mois 10
############################################################################
myMonthNumber = 10
dfAMThisMonthPV = getMyDistribution(myPageViews=dfAMPageViews,
myArticles=myArticles,
myNumPeriode=myMonthNumber,
myNbrOfDays=30,
myLastDate=lastDate,
myTestType="AM")
#test de normalité shapiro wilk
dfAMThisMonthPVDropNa = dfAMThisMonthPV.dropna() #
myW, myPValeur = stats.shapiro(dfAMThisMonthPVDropNa['ThisPeriodPV'].values)
myPValeur #normalité rejeté
sns.set() #paramètres esthétiques ressemble à ggplot par défaut.
fig, ax = plt.subplots() #un seul plot
sns.distplot(dfAMThisMonthPVDropNa['ThisPeriodPV'].values, bins=30, kde=False, rug=True)
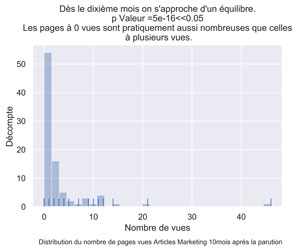
ax.set(xlabel="Nombre de vues", ylabel='Décompte',
title="Dès le dixième mois on s'approche d'un équilibre.\n p Valeur =" + str(round(myPValeur,16)) +
"<<0.05 \n Les pages à 0 vues sont pratiquement aussi nombreuses que celles \n à plusieurs vues.")
fig.text(.9,-.05,"Distribution du nombre de pages vues Articles Marketing " + str(myMonthNumber) + "mois après la parution",
fontsize=9, ha="right")
#plt.show()
fig.savefig("Dist-PV-AM-Mois-"+str(myMonthNumber)+".png", bbox_inches="tight", dpi=600)
POUR LE MOIS 40 :
############################################################################
# Pour le mois 40
############################################################################
myMonthNumber = 40
dfAMThisMonthPV = getMyDistribution(myPageViews=dfAMPageViews,
myArticles=myArticles,
myNumPeriode=myMonthNumber,
myNbrOfDays=30,
myLastDate=lastDate,
myTestType="AM")
#test de normalité shapiro wilk
dfAMThisMonthPVDropNa = dfAMThisMonthPV.dropna() #
myW, myPValeur = stats.shapiro(dfAMThisMonthPVDropNa['ThisPeriodPV'].values)
myPValeur #normalité rejeté
sns.set() #paramètres esthétiques ressemble à ggplot par défaut.
fig, ax = plt.subplots() #un seul plot
sns.distplot(dfAMThisMonthPVDropNa['ThisPeriodPV'].values, bins=30, kde=False, rug=True)
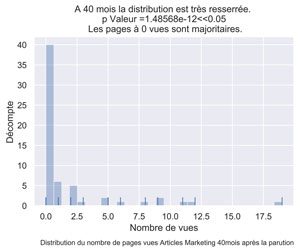
ax.set(xlabel="Nombre de vues", ylabel='Décompte',
title="A 40 mois la distribution est très resserrée. \n p Valeur =" + str(round(myPValeur,17)) +
"<<0.05 \n Les pages à 0 vues sont majoritaires.")
fig.text(.9,-.05,"Distribution du nombre de pages vues Articles Marketing " + str(myMonthNumber) + "mois après la parution",
fontsize=9, ha="right")
#plt.show()
fig.savefig("Dist-PV-AM-Mois-"+str(myMonthNumber)+".png", bbox_inches="tight", dpi=600)
Pour nos données, on constate visuellement que dès le 10ème mois, les pages articles marketing n’apportent plus de trafic.
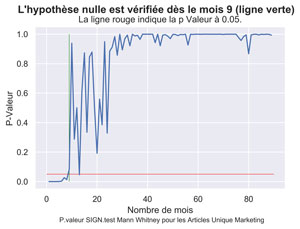
Nous allons vérifier cela statistiquement avec un SIGN.test. En Python nous utiliserons le test de rang de Mann-Whitney qui permet de tester une alternative « supérieur ». Ici on teste l’hypothèse H0 trafic = 0 pages vues et H1 trafic > 0.
Significativité du trafic « articles marketing » dans les mois suivants la publication.
Utilisation du test de Mann-Whitney pour tester la significativité du trafic « Articles Marketing »
#############################################################################################
# Utilisation du SIGN.test pour tester la significativité des distributions.
#############################################################################################
#initialisation :
#Pour enregistrer les données du test pour toutes les distributions
dfAMPValue = pd.DataFrame(columns= ['pvalue', 'statistic',
'myNotNas', 'myNotNull','myMedian'])
myAMMd = 0.01 #médiane de l'hypothèse nulle : 0 ne marche pas
#-> à mon avis le test est que la médiane soit inférieure
#à cette valeur donc < 0 ne donne rien alors que < 0.01
#détecte les 0.
#Rem cela fonctionne pareil avec SAS, R et Python
myAMCl = 0.95 #niveau de confiance souhaité. non utilisé ici
myLastMonth = 90 #dernier mois à investiguer 7,5 années
lastDate = dfPageViews.iloc[len(dfPageViews.index)-1]['date']
#x=1
for x in range(1,myLastMonth):
dfAMThisMonthPV = getMyDistribution(myPageViews=dfAMPageViews,
myArticles=myArticles,
myNumPeriode=x,
myNbrOfDays=30,
myLastDate=lastDate,
myTestType="AM")
dfAMThisMonthPVDropNa = dfAMThisMonthPV.dropna()
#on compare par rapport à une distribution presque à zéro : 0.01
zeroData = pd.DataFrame(myAMMd, index=np.arange(len(dfAMThisMonthPVDropNa)), columns=['ThisPeriodPV'])
##### Différents essai de stats
#statistic, pvalue = sp.stats.wilcoxon(x=dfAMThisMonthPVDropNa['ThisPeriodPV'].values, y=None, zero_method='wilcox', correction=False)
#statistic, pvalue = sp.stats.ranksums(x=dfAMThisMonthPVDropNa['ThisPeriodPV'].values, y=zeroData['ThisPeriodPV'].values)
#mannwhitneyu permet d'avoir une alternative "Greater"
statistic, pvalue = sp.stats.mannwhitneyu(x=dfAMThisMonthPVDropNa['ThisPeriodPV'].values, y=zeroData['ThisPeriodPV'].values, alternative='greater')
#statistic *100
statistic = statistic/100
myNotNas = len(dfAMThisMonthPVDropNa) #nombre d'observations non NaN
myNotNull = (dfAMThisMonthPVDropNa['ThisPeriodPV']>0).sum() #nombre d'observations non nulle
dfAMPValue = dfAMPValue.append({'pvalue': pvalue, 'statistic':statistic, 'myNotNas':myNotNas
, 'myNotNull':myNotNull}, ignore_index=True)
dfAMPValue.index #pour l'axe des x.
myfirstMonthPValueUpper = dfAMPValue.index.get_loc(dfAMPValue.index[dfAMPValue['pvalue'] > 0.05][0]) + 1
#graphique
sns.set() #paramètres esthétiques ressemble à ggplot par défaut.
fig, ax = plt.subplots() #un seul plot
sns.lineplot(x=dfAMPValue.index.values+1, y=dfAMPValue.pvalue)
plt.vlines(x = myfirstMonthPValueUpper, ymin = 0, ymax = 1, color = 'green', linewidth=0.5)
plt.hlines(y = 0.05, xmin = 0, xmax = 90, color = 'red', linewidth=0.5)
fig.suptitle("L'hypothèse nulle est vérifiée dès le mois "+ str(myfirstMonthPValueUpper) + " (ligne verte)", fontsize=14, fontweight='bold')
ax.set(xlabel='Nombre de mois', ylabel='P-Valeur',
title='La ligne rouge indique la p Valeur à 0.05.')
fig.text(.3,-.03,"P.valeur SIGN.test Mann Withney pour les Articles Marketing",
fontsize=9)
#plt.show()
fig.savefig("AM-SIGN-Test-P-Value.png", bbox_inches="tight", dpi=600)
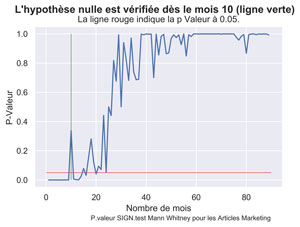
Comparatif Statistique vs taille de l’échantillon
#comparons la statistique càd ici les pages avec vues > 0
#vs la taille de l'échantillon donnés par myNotNas, et la moitié de ce dernier
sns.set() #paramètres esthétiques ressemble à ggplot par défaut.
fig, ax = plt.subplots() #un seul plot
sns.lineplot(x=dfAMPValue.index.values+1, y=dfAMPValue.statistic, color="blue")
sns.lineplot(x=dfAMPValue.index.values+1, y=dfAMPValue.myNotNas, color="red")
sns.lineplot(x=dfAMPValue.index.values+1, y=dfAMPValue.myNotNas/2, color="black")
fig.suptitle("Le nombre de pages avec vues > 0 baisse plus vite que la taille de l'échantillon.\n La courbe bleu s'approche rapidement de la ligne noire \n qui représente la moitié de l'échantillon.", fontsize=10, fontweight='bold')
ax.set(xlabel="Nombre de mois", ylabel="Pages Vues > 0 (bleu) / Taille échantillon (rouge)",
title="")
fig.text(.2,-.03,"Evolution mensuelle du Nbr de pages vues > 0 vs Taille échantillon", fontsize=9)
#plt.show()
fig.savefig("AM-sup0-SampleSize.png", bbox_inches="tight", dpi=600)
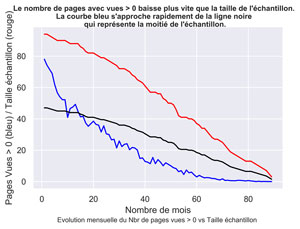
Autre méthode : calcul de l’intervalle de confiance pour une proportion
Voir dans l’article avec R pour les explications.
##################################################################################
# vérifions en calculant l'intervalle de confiance à 95% sur une proportion
# avec les données observées.
dfAMPValue['prop'] = dfAMPValue.apply(lambda row: row.myNotNull / row.myNotNas, axis=1) #proportion
#Intervalle de confiance à 95%
dfAMPValue['confIntProportion'] = dfAMPValue.apply(lambda row: 1.96 * math.sqrt((row.prop*(1-row.prop))/row.myNotNas) , axis=1)
#borne inférieure
dfAMPValue['propCIinf'] = dfAMPValue.apply(lambda row: row.prop - row.confIntProportion, axis=1)
#borne superieure
dfAMPValue['propCIsup'] = dfAMPValue.apply(lambda row: row.prop + row.confIntProportion, axis=1)
#Première valeur dela borne inférieure sous 0.5
firstpropCIinfUnder = dfAMPValue.index.get_loc(dfAMPValue.index[dfAMPValue['propCIinf'] <= 0.5][0]) + 1
###################################################################################
sns.set() #paramètres esthétiques ressemble à ggplot par défaut.
fig, ax = plt.subplots() #un seul plot
sns.lineplot(x=dfAMPValue.index.values+1, y=dfAMPValue.prop, color="black")
sns.lineplot(x=dfAMPValue.index.values+1, y=dfAMPValue.propCIsup, color="blue")
sns.lineplot(x=dfAMPValue.index.values+1, y=dfAMPValue.propCIinf, color="blue")
plt.vlines(x = firstpropCIinfUnder, ymin = 0, ymax = 1, color = 'green', linewidth=0.5)
plt.hlines(y = 0.5, xmin = 0, xmax = 90, color = 'red', linewidth=0.5)
fig.suptitle("L'hypothèse nulle est vérifiée dès le mois " + str(firstpropCIinfUnder) , fontsize=14, fontweight='bold')
ax.set(xlabel="Nombre de mois", ylabel="proportion avec intervalle de confiance (bleu) ",
title="La valeur inférieure de l'IC passe sous la barre des 0.5")
fig.text(.2,-.03,"Proportion de pages vues > 0 pour chaque distribution mensuelle ", fontsize=9)
#plt.show()
fig.savefig("AM-PropPVsup1.png", bbox_inches="tight", dpi=600)
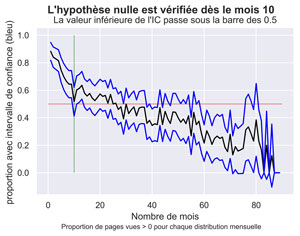
On obtient le même résultat qu’avec le test de Mann-Whitney, soit le mois 10.
Résultats pour les valeurs lissées
Pour les valeurs lissés nous allons utiliser une fonction Loess développée par James D. Triveri : http://www.jtrive.com/loess-nonparametric-scatterplot-smoothing-in-python.html.
###################################################################################
#en Lissage Loess
#Calcul Valeurs lissées
###################################################################
# Fonction Loess récupérée sur le Net : ( James Triveri)
# http://www.jtrive.com/loess-nonparametric-scatterplot-smoothing-in-python.html
####################################################################################
"""
Local Regression (LOESS) estimation routine.
"""
import numpy as np
import pandas as pd
import scipy
def loc_eval(x, b):
"""
Evaluate `x` using locally-weighted regression parameters.
Degree of polynomial used in loess is inferred from b. `x`
is assumed to be a scalar.
"""
loc_est = 0
for i in enumerate(b): loc_est+=i[1]*(x**i[0])
return(loc_est)
def loess(xvals, yvals, alpha, poly_degree=1):
"""
Perform locally-weighted regression on xvals & yvals.
Variables used inside `loess` function:
n => number of data points in xvals
m => nbr of LOESS evaluation points
q => number of data points used for each
locally-weighted regression
v => x-value locations for evaluating LOESS
locsDF => contains local regression details for each
location v
evalDF => contains actual LOESS output for each v
X => n-by-(poly_degree+1) design matrix
W => n-by-n diagonal weight matrix for each
local regression
y => yvals
b => local regression coefficient estimates.
b = `(X^T*W*X)^-1*X^T*W*y`. Note that `@`
replaces `np.dot` in recent numpy versions.
local_est => response for local regression
"""
# Sort dataset by xvals.
all_data = sorted(zip(xvals, yvals), key=lambda x: x[0])
xvals, yvals = zip(*all_data)
locsDF = pd.DataFrame(
columns=[
'loc','x','weights','v','y','raw_dists',
'scale_factor','scaled_dists'
])
evalDF = pd.DataFrame(
columns=[
'loc','est','b','v','g'
])
n = len(xvals)
m = n + 1
q = int(np.floor(n * alpha) if alpha <= 1.0 else n)
avg_interval = ((max(xvals)-min(xvals))/len(xvals))
v_lb = max(0,min(xvals)-(.5*avg_interval))
v_ub = (max(xvals)+(.5*avg_interval))
v = enumerate(np.linspace(start=v_lb, stop=v_ub, num=m), start=1)
#print('liste v=', list(v))
# Generate design matrix based on poly_degree.
xcols = [np.ones_like(xvals)]
for j in range(1, (poly_degree + 1)):
xcols.append([i ** j for i in xvals])
X = np.vstack(xcols).T
for i in v:
print('i=', i) #pour voir à quelle vitesse cela défile.
iterpos = i[0]
iterval = i[1]
# Determine q-nearest xvals to iterval.
iterdists = sorted([(j, np.abs(j-iterval)) \
for j in xvals], key=lambda x: x[1])
_, raw_dists = zip(*iterdists)
# Scale local observations by qth-nearest raw_dist.
scale_fact = raw_dists[q-1]
scaled_dists = [(j[0],(j[1]/scale_fact)) for j in iterdists]
weights = [(j[0],((1-np.abs(j[1]**3))**3 \
if j[1]<=1 else 0)) for j in scaled_dists]
# Remove xvals from each tuple:
_, weights = zip(*sorted(weights, key=lambda x: x[0]))
_, raw_dists = zip(*sorted(iterdists, key=lambda x: x[0]))
_, scaled_dists = zip(*sorted(scaled_dists,key=lambda x: x[0]))
iterDF1 = pd.DataFrame({
'loc' :iterpos,
'x' :xvals,
'v' :iterval,
'weights' :weights,
'y' :yvals,
'raw_dists' :raw_dists,
'scale_fact' :scale_fact,
'scaled_dists':scaled_dists
})
locsDF = pd.concat([locsDF, iterDF1])
W = np.diag(weights)
y = yvals
b = np.linalg.inv(X.T @ W @ X) @ (X.T @ W @ y)
local_est = loc_eval(iterval, b)
iterDF2 = pd.DataFrame({
'loc':[iterpos],
'b' :[b],
'v' :[iterval],
'g' :[local_est]
})
evalDF = pd.concat([evalDF, iterDF2])
# Reset indicies for returned DataFrames.
locsDF.reset_index(inplace=True)
locsDF.drop('index', axis=1, inplace=True)
locsDF['est'] = 0; evalDF['est'] = 0
locsDF = locsDF[['loc','est','v','x','y','raw_dists',
'scale_fact','scaled_dists','weights']]
# Reset index for evalDF.
evalDF.reset_index(inplace=True)
evalDF.drop('index', axis=1, inplace=True)
evalDF = evalDF[['loc','est', 'v', 'b', 'g']]
return(locsDF, evalDF)
######################################################################"
Effectuons nos calculs et créons le graphique :
#Calculs
regsDF, myAMLoess = loess(dfAMPValue.index.values,dfAMPValue.prop.values, alpha=.34, poly_degree=1)
myAMLoess.g #<- valeurs lissées.
# valeurs lissées inférieures :
myAMLoess['conf.int.inf'] = myAMLoess.apply(lambda row: row.g - 1.96*stats.sem(myAMLoess.g) , axis=1)
#Première valeur de la borne inférieure de l'IC lissée sous la barre des 0.5 i.e mediane = 0
firstAMLoessCIFUnder = myAMLoess.index.get_loc(myAMLoess.index[myAMLoess['conf.int.inf'] <= 0.5][0]) + 1
# valeurs lissées supérieures :
myAMLoess['conf.int.sup'] = myAMLoess.apply(lambda row: row.g + 1.96*stats.sem(myAMLoess.g) , axis=1)
###################################################################################
sns.set() #paramètres esthétiques ressemble à ggplot par défaut.
fig, ax = plt.subplots() #un seul plot
sns.lineplot(x=dfAMPValue.index.values+1, y=dfAMPValue.prop, color="black")
sns.lineplot(x=myAMLoess.index.values+1, y=myAMLoess["conf.int.sup"], color="grey", alpha=0.5)
sns.lineplot(x=myAMLoess.index.values+1, y=myAMLoess.g, color="blue")
sns.lineplot(x=myAMLoess.index.values+1, y=myAMLoess["conf.int.inf"], color="grey", alpha=0.5)
plt.vlines(x = firstAMLoessCIFUnder, ymin = 0, ymax = 1, color = 'green', linewidth=0.5)
plt.hlines(y = 0.5, xmin = 0, xmax = 90, color = 'red', linewidth=0.5)
fig.suptitle("L'hypothèse nulle est vérifiée au mois " + str(firstAMLoessCIFUnder) , fontsize=14, fontweight='bold')
ax.set(xlabel="Nombre de mois", ylabel="proportion lissée (bleu)",
title="La valeur inférieure de l'IC passe sous la barre des 0.5 (rouge)")
fig.text(.2,-.03,"Proportion lissée de pages vues > 0 pour chaque distribution mensuelle", fontsize=9)
#plt.show()
fig.savefig("AM-PropPVsup1-Loess.png", bbox_inches="tight", dpi=600)
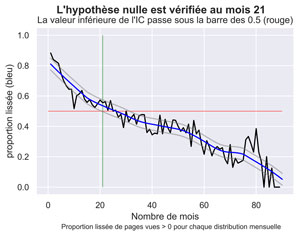
Significativité du trafic « direct marketing » dans les mois suivants la publication.
On répète l’opération précédente pour les pages « Direct Marketing ». Il s’agit des pages dont l’entrée s’est faite via une page article marketing.
Utilisation du test de Mann-Whitney pour tester la significativité du trafic « DIRECT Marketing »
##########################################################################
# Restreignons l'investigation aux pages "Marketing" visitées uniquement
# suite à une entrée sur une page "Marketing" (la même ou une autre)
##########################################################################
#Verifs ############
dfPageViews.info()
myArticles.info()
pattern = '|'.join(myArticles['pagePath'])
#on enlève les pagePath
indexLandingPagePathToKeep = dfPageViews[(dfPageViews.landingPagePath.str.contains(pat=pattern,regex=True)==True)].index
dfDMPageViews = dfPageViews.iloc[indexLandingPagePathToKeep]
dfDMPageViews.reset_index(inplace=True, drop=True) #on reindexe
dfDMPageViews.count() #28553 obs.
###############################################################################
# Sauvegarde en csv si besoin
dfDMPageViews.to_csv("dfDMPageViews.csv", sep=";", index=False) #séparateur ;
###############################################################################
#Preparation des données pour graphique de pvalue
dfDMPValue = pd.DataFrame(columns= ['pvalue', 'statistic',
'myNotNas', 'myNotNull','myMedian'])
myDMMd = 0.01 #médiane de l'hypothèse nulle : 0 ne marche pas
myLastMonth = 90 #dernier mois à investiguer 7,5 années
lastDate = dfDMPageViews.iloc[len(dfDMPageViews.index)-1]['date']
#x=1
for x in range(1,myLastMonth):
dfDMThisMonthPV = getMyDistribution(myPageViews=dfDMPageViews,
myArticles=myArticles,
myNumPeriode=x,
myNbrOfDays=30,
myLastDate=lastDate,
myTestType="AM")
dfDMThisMonthPVDropNa = dfDMThisMonthPV.dropna()
#on compare par rapport à une distribution presque à zéro : 0.01
zeroData = pd.DataFrame(myDMMd, index=np.arange(len(dfDMThisMonthPVDropNa)), columns=['ThisPeriodPV'])
#mannwhitneyu permet d'avoir une alternative "Greater"
statistic, pvalue = sp.stats.mannwhitneyu(x=dfDMThisMonthPVDropNa['ThisPeriodPV'].values, y=zeroData['ThisPeriodPV'].values, alternative='greater')
#statistic est *100 ne sais pas pourquoi
statistic = statistic/100
myNotNas = len(dfDMThisMonthPVDropNa) #nombre d'observations non NaN
myNotNull = (dfDMThisMonthPVDropNa['ThisPeriodPV']>0).sum() #nombre d'observations non nulle
dfDMPValue = dfDMPValue.append({'pvalue': pvalue, 'statistic':statistic, 'myNotNas':myNotNas
, 'myNotNull':myNotNull}, ignore_index=True)
dfDMPValue.index #pour l'axe des x.
myfirstMonthPValueUpper = dfDMPValue.index.get_loc(dfDMPValue.index[dfDMPValue['pvalue'] > 0.05][0]) + 1
#graphique
sns.set() #paramètres esthétiques ressemble à ggplot par défaut.
fig, ax = plt.subplots() #un seul plot
sns.lineplot(x=dfDMPValue.index.values+1, y=dfDMPValue.pvalue)
plt.vlines(x = myfirstMonthPValueUpper, ymin = 0, ymax = 1, color = 'green', linewidth=0.5)
plt.hlines(y = 0.05, xmin = 0, xmax = 90, color = 'red', linewidth=0.5)
fig.suptitle("L'hypothèse nulle est vérifiée dès le mois "+ str(myfirstMonthPValueUpper) + " (ligne verte)", fontsize=14, fontweight='bold')
ax.set(xlabel='Nombre de mois', ylabel='P-Valeur',
title='La ligne rouge indique la p Valeur à 0.05.')
fig.text(.2,-.03,"P.valeur SIGN.test Mann Whitney pour les Articles Direct Marketing",
fontsize=9)
#plt.show()
fig.savefig("DM-SIGN-Test-P-Value.png", bbox_inches="tight", dpi=600)
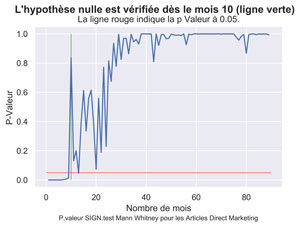
Comparatif Articles Marketing vs DIRECT MARKETING LISSE
#########################################################
## Comparatifs AM MD lissés
#on aura besoin de la proportion ensuite
dfDMPValue['prop'] = dfDMPValue.apply(lambda row: row.myNotNull / row.myNotNas, axis=1) #proportion
#Calcul Valeurs lissées pour Direct Marketing
regsDF, myDMLoess = loess(dfDMPValue.index.values,dfDMPValue.prop.values, alpha=.34, poly_degree=1)
myDMLoess.g #<- valeurs lissées.
# valeurs lissées inférieures :
myDMLoess['conf.int.inf'] = myDMLoess.apply(lambda row: row.g - 1.96*stats.sem(myDMLoess.g) , axis=1)
#Première valeur de la borne inférieure de l'IC lissée sous la barre des 0.5 i.e mediane = 0
firstDMLoessCIFUnder = myDMLoess.index.get_loc(myDMLoess.index[myDMLoess['conf.int.inf'] <= 0.5][0]) + 1
# valeurs lissées supérieures :
myDMLoess['conf.int.sup'] = myDMLoess.apply(lambda row: row.g + 1.96*stats.sem(myDMLoess.g) , axis=1)
###################################################################################
sns.set() #paramètres esthétiques ressemble à ggplot par défaut.
fig, ax = plt.subplots() #un seul plot
sns.lineplot(x=dfAMPValue.index.values+1, y=dfAMPValue.prop, color="blue")
sns.lineplot(x=myAMLoess.index.values+1, y=myAMLoess["conf.int.sup"], color="grey", alpha=0.5)
sns.lineplot(x=myAMLoess.index.values+1, y=myAMLoess.g, color="blue")
sns.lineplot(x=myAMLoess.index.values+1, y=myAMLoess["conf.int.inf"], color="grey", alpha=0.5)
sns.lineplot(x=dfDMPValue.index.values+1, y=dfDMPValue.prop, color="red")
sns.lineplot(x=myDMLoess.index.values+1, y=myDMLoess["conf.int.sup"], color="grey", alpha=0.5)
sns.lineplot(x=myDMLoess.index.values+1, y=myDMLoess.g, color="red")
sns.lineplot(x=myDMLoess.index.values+1, y=myDMLoess["conf.int.inf"], color="grey", alpha=0.5)
plt.vlines(x = firstDMLoessCIFUnder, ymin = 0, ymax = 1, color = 'green', linewidth=0.5)
plt.hlines(y = 0.5, xmin = 0, xmax = 90, color = 'red', linewidth=0.5)
fig.suptitle("L'hypothèse nulle pour Direct Marketing est vérifiée au mois " + str(firstDMLoessCIFUnder) , fontsize=14, fontweight='bold')
ax.set(xlabel="Nombre de mois", ylabel="proportion lissée - AM: bleu, DM : rouge ",
title="La valeur inférieure DM de l'IC passe sous la barre des 0.5 (rouge)")
fig.text(.2,-.05,"Proportion lissée Articles Marketing et Direct Marketing de pages vues > 0 \n pour chaque distribution mensuelle", fontsize=9)
#plt.show()
fig.savefig("DM-AM-PropPVsup1-Loess.png", bbox_inches="tight", dpi=600)
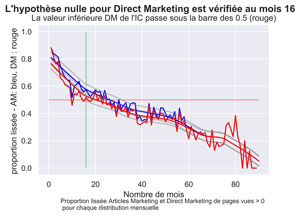
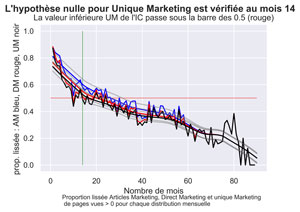
Significativité du trafic « unique marketing » dans les mois suivants la publication.
Il s’agit des pages « Marketing » visitées uniquement suite à une entrée sur la même page « Marketing ».
#################################################################
# Restreignons encore l'investigation aux pages visités
# uniquement suite à une entrée sur la même page "Marketing"
# UM : Unique Marketing
#################################################################
#on garde uniquement les landingPagePath = pagePath
dfUMPageViews = dfDMPageViews[(dfDMPageViews.landingPagePath == dfDMPageViews.pagePath)]
dfUMPageViews.reset_index(inplace=True, drop=True) #on reindexe
dfUMPageViews.count() #21214 obs.
#Preparation des données pour graphique de pvalue
dfUMPValue = pd.DataFrame(columns= ['pvalue', 'statistic',
'myNotNas', 'myNotNull'])
myUMMd = 0.01 #médiane de l'hypothèse nulle : 0 ne marche pas avec R
myLastMonth = 90 #dernier mois à investiguer 7,5 années
lastDate = dfUMPageViews.iloc[len(dfUMPageViews.index)-1]['date']
#x=1
for x in range(1,myLastMonth):
dfUMThisMonthPV = getMyDistribution(myPageViews=dfUMPageViews,
myArticles=myArticles,
myNumPeriode=x,
myNbrOfDays=30,
myLastDate=lastDate,
myTestType="AM")
dfUMThisMonthPVDropNa = dfUMThisMonthPV.dropna()
#on compare par rapport à une distribution presque à zéro : 0.01
zeroData = pd.DataFrame(myUMMd, index=np.arange(len(dfUMThisMonthPVDropNa)), columns=['ThisPeriodPV'])
#mannwhitneyu permet d'avoir une alternative "Greater"
statistic, pvalue = sp.stats.mannwhitneyu(x=dfUMThisMonthPVDropNa['ThisPeriodPV'].values, y=zeroData['ThisPeriodPV'].values, alternative='greater')
#statistic est *100 ne sais pas pourquoi
statistic = statistic/100
myNotNas = len(dfUMThisMonthPVDropNa) #nombre d'observations non NaN
myNotNull = (dfUMThisMonthPVDropNa['ThisPeriodPV']>0).sum() #nombre d'observations non nulle
dfUMPValue = dfUMPValue.append({'pvalue': pvalue, 'statistic':statistic, 'myNotNas':myNotNas
, 'myNotNull':myNotNull}, ignore_index=True)
dfUMPValue.index #pour l'axe des x.
myfirstMonthPValueUpper = dfUMPValue.index.get_loc(dfUMPValue.index[dfUMPValue['pvalue'] > 0.05][0]) + 1
#graphique
sns.set() #paramètres esthétiques ressemble à ggplot par défaut.
fig, ax = plt.subplots() #un seul plot
sns.lineplot(x=dfUMPValue.index.values+1, y=dfUMPValue.pvalue)
plt.vlines(x = myfirstMonthPValueUpper, ymin = 0, ymax = 1, color = 'green', linewidth=0.5)
plt.hlines(y = 0.05, xmin = 0, xmax = 90, color = 'red', linewidth=0.5)
fig.suptitle("L'hypothèse nulle est vérifiée dès le mois "+ str(myfirstMonthPValueUpper) + " (ligne verte)", fontsize=14, fontweight='bold')
ax.set(xlabel='Nombre de mois', ylabel='P-Valeur',
title='La ligne rouge indique la p Valeur à 0.05.')
fig.text(.2,-.03,"P.valeur SIGN.test Mann Whitney pour les Articles Unique Marketing",
fontsize=9)
#plt.show()
fig.savefig("UM-SIGN-Test-P-Value.png", bbox_inches="tight", dpi=600)
Proportions lissées, comparatif ArtIcles Marketing, Direct Marketing ET UNIQUE MARKETING
#################################################################
## Comparatifs AM DM UM
#################################################################
#Calcul Valeurs lissées pour Unique Marketing
#on aura besoin de la proportion ensuite
dfUMPValue['prop'] = dfUMPValue.apply(lambda row: row.myNotNull / row.myNotNas, axis=1) #proportion
#Calcul Valeurs lissées pour Direct Marketing
regsDF, myUMLoess = loess(dfUMPValue.index.values,dfUMPValue.prop.values, alpha=.34, poly_degree=1)
myUMLoess.g #<- valeurs lissées.
# valeurs lissées inférieures :
myUMLoess['conf.int.inf'] = myUMLoess.apply(lambda row: row.g - 1.96*stats.sem(myUMLoess.g) , axis=1)
#Première valeur de la borne inférieure de l'IC lissée sous la barre des 0.5 i.e mediane = 0
firstUMLoessCIFUnder = myUMLoess.index.get_loc(myUMLoess.index[myUMLoess['conf.int.inf'] <= 0.5][0]) + 1
# valeurs lissées supérieures :
myUMLoess['conf.int.sup'] = myUMLoess.apply(lambda row: row.g + 1.96*stats.sem(myUMLoess.g) , axis=1)
###################################################################################
sns.set() #paramètres esthétiques ressemble à ggplot par défaut.
fig, ax = plt.subplots() #un seul plot
sns.lineplot(x=dfAMPValue.index.values+1, y=dfAMPValue.prop, color="blue")
sns.lineplot(x=myAMLoess.index.values+1, y=myAMLoess["conf.int.sup"], color="grey", alpha=0.5)
sns.lineplot(x=myAMLoess.index.values+1, y=myAMLoess.g, color="blue")
sns.lineplot(x=myAMLoess.index.values+1, y=myAMLoess["conf.int.inf"], color="grey", alpha=0.5)
sns.lineplot(x=dfDMPValue.index.values+1, y=dfDMPValue.prop, color="red")
sns.lineplot(x=myDMLoess.index.values+1, y=myDMLoess["conf.int.sup"], color="grey", alpha=0.5)
sns.lineplot(x=myDMLoess.index.values+1, y=myDMLoess.g, color="red")
sns.lineplot(x=myDMLoess.index.values+1, y=myDMLoess["conf.int.inf"], color="grey", alpha=0.5)
sns.lineplot(x=dfUMPValue.index.values+1, y=dfUMPValue.prop, color="black")
sns.lineplot(x=myUMLoess.index.values+1, y=myUMLoess["conf.int.sup"], color="grey", alpha=0.5)
sns.lineplot(x=myUMLoess.index.values+1, y=myUMLoess.g, color="black")
sns.lineplot(x=myUMLoess.index.values+1, y=myUMLoess["conf.int.inf"], color="grey", alpha=0.5)
plt.vlines(x = firstUMLoessCIFUnder, ymin = 0, ymax = 1, color = 'green', linewidth=0.5)
plt.hlines(y = 0.5, xmin = 0, xmax = 90, color = 'red', linewidth=0.5)
fig.suptitle("L'hypothèse nulle pour Unique Marketing est vérifiée au mois " + str(firstUMLoessCIFUnder) , fontsize=14, fontweight='bold')
ax.set(xlabel="Nombre de mois", ylabel="prop. lissée : AM bleu, DM rouge, UM noir ",
title="La valeur inférieure UM de l'IC passe sous la barre des 0.5 (rouge)")
fig.text(.2,-.05,"Proportion lissée Articles Marketing, Direct Marketing et unique Marketing \n de pages vues > 0 pour chaque distribution mensuelle", fontsize=9)
#plt.show()
fig.savefig("UM-DM-AM-PropPVsup1-Loess.png", bbox_inches="tight", dpi=600)
##########################################################################
# MERCI pour votre attention !
##########################################################################
#on reste dans l'IDE
#if __name__ == '__main__':
# main()
Conclusion
Le test de Mann Whitney et la fonction Loess utilisée en Python donnent des résultats légèrement différents de ceux que nous avons obtenus avec SIGN.test et la fonction Loess de R.
Ici l’efficacité se situe entre 9 et 21 mois selon que l’on a une vision restrictive et/ou que l’on travaille avec des données brutes ou non.
Comme, nous l’indiquions précédemment, ces résultats ne valent que pour un seul site et un groupe d’articles et il serait intéressant de refaire cette démarche pour d’autres sites et ou pages.
N’hésitez pas à communiquer vos résultats en commentaires.
A Bientôt,
Pierre